अध्याय 07 बुनियादी प्रक्रियाएँ
7.1 डीएनए अनुवांशिक पदार्थ के रूप में
आपने पिछले अध्याय में पढ़ा है कि लक्षण या विशेषताएँ जीनों के माध्यम से माता-पिता से संतान तक आती हैं। आप यह भी जानते हैं कि ये जीन गुणसूत्रों पर मौजूद होते हैं जो न्यूक्लिक अम्ल और प्रोटीन से बने होते हैं। हालांकि, किसी लक्षण के अभिव्यक्ति के लिए उत्तरदायी जीन की प्रकृति को समझना वैज्ञानिक समुदाय के सामने सबसे बड़ी चुनौतियों में से एक था। इस प्रश्न का उत्तर कुछ प्रयोगात्मक साक्ष्यों के बाद मिला जिनसे पता चला कि डीऑक्सीराइबोन्यूक्लिक अम्ल (डीएनए) किसी भी जीव की विशेषता या लक्षण निर्धारित करता है, कुछ कुछ वायरसों को छोड़कर।
डीएनए की खोज का श्रेय जोहान फ्रिडरिक मीशर को जाता है, जिसने पहली बार पस की कोशिकाओं के नाभिक से एक अम्लीय पदार्थ अलग किया और उसे न्यूक्लिन नाम दिया जिसमें डीएनए और प्रोटीन होते हैं। गुणसूत्र और नाभिक में उपस्थिति के कारण ये दो रासायनिक घटक; न्यूक्लिक अम्ल (मुख्यतः डीएनए) और प्रोटीन संभावित उम्मीदवार बन गए कि ये अनुवांशिक पदार्थ हो सकते हैं। फिर भी, अनुवांशिक पदार्थ की प्रकृति लंबे समय तक अज्ञात रही। धीरे-धीरे, विभिन्न अन्वेषकों द्वारा सूक्ष्मजीवों के साथ किए गए प्रयोगों ने ऐसे परिणाम दिए जिन्होंने डीएनए को अनुवांशिक पदार्थ के रूप में पक्ष में साक्ष्य प्रदान किए।
7.1.1 रूपांतरण सिद्धांत की खोज
1928 में, एक ब्रिटिश चिकित्सा अधिकारी, फ्रेडरिक ग्रिफिथ ने स्तनधारियों में बैक्टीरियम स्ट्रेप्टोकोकस न्यूमोनिया (जिसे डिप्लोकोकस न्यूमोनिया भी कहा जाता है) के कारण होने वाले न्यूमोनिया के खिलाफ एक वैक्सीन विकसित करने की प्रक्रिया में एक प्रेक्षण किया, जो मनुष्यों में न्यूमोनिया का कारण बनता है और चूहों में सामान्यतः घातक होता है। उसने इस बैक्टीरिया की दो भिन्न उपभेदों (किस्मों) की पहचान की, अर्थात् विषाणुजनक (रोग उत्पन्न करने वाली) जिसके चारों ओर एक पॉलिसैकेराइड कैप्सूल होता है और अविषाणुजनक (हानिरहित)। विषाणुजनक उपभेद में, प्रत्येक बैक्टीरियम को एक पॉलिसैकेराइड कैप्सूल घेरे रहता है जिसके कारण जब बैक्टीरियल कॉलोनी को एक ऐगर प्लेट पर उगाया जाता है तो वह चिकनी दिखती है और इसे चिकनी उपभेद (S) कहा जाता है। अविषाणुजनक उपभेद में पॉलिसैकेराइड आवरण नहीं होता और ये खुरदुरी दिखने वाली कॉलोनी उत्पन्न करते हैं और इन्हें खुरदुरी उपभेद (R) कहा जाता है। S प्रकार के बैक्टीरिया न्यूमोनिया उत्पन्न करके चूहों को मार देते हैं।
ग्रिफिथ ने S और R प्रकार के बैक्टीरिया के साथ एक श्रृंखला प्रयोग किए (चित्र 7.1)। जब उसने जीवित S बैक्टीरिया को चूहों में इंजेक्ट किया, तो चूहों को न्यूमोनिया हुआ और वे मर गए। हालांकि, जब उसने चूहों को R प्रकार के बैक्टीरिया से संक्रमित किया तो चूहों में कोई बुरा प्रभाव नहीं दिखा। इन दो प्रयोगों के परिणामों ने पुष्टि की कि S प्रकार के बैक्टीरिया में मौजूद पॉलिसैकेराइड आवरण स्पष्ट रूप से विषाणुजनकता के लिए आवश्यक था।
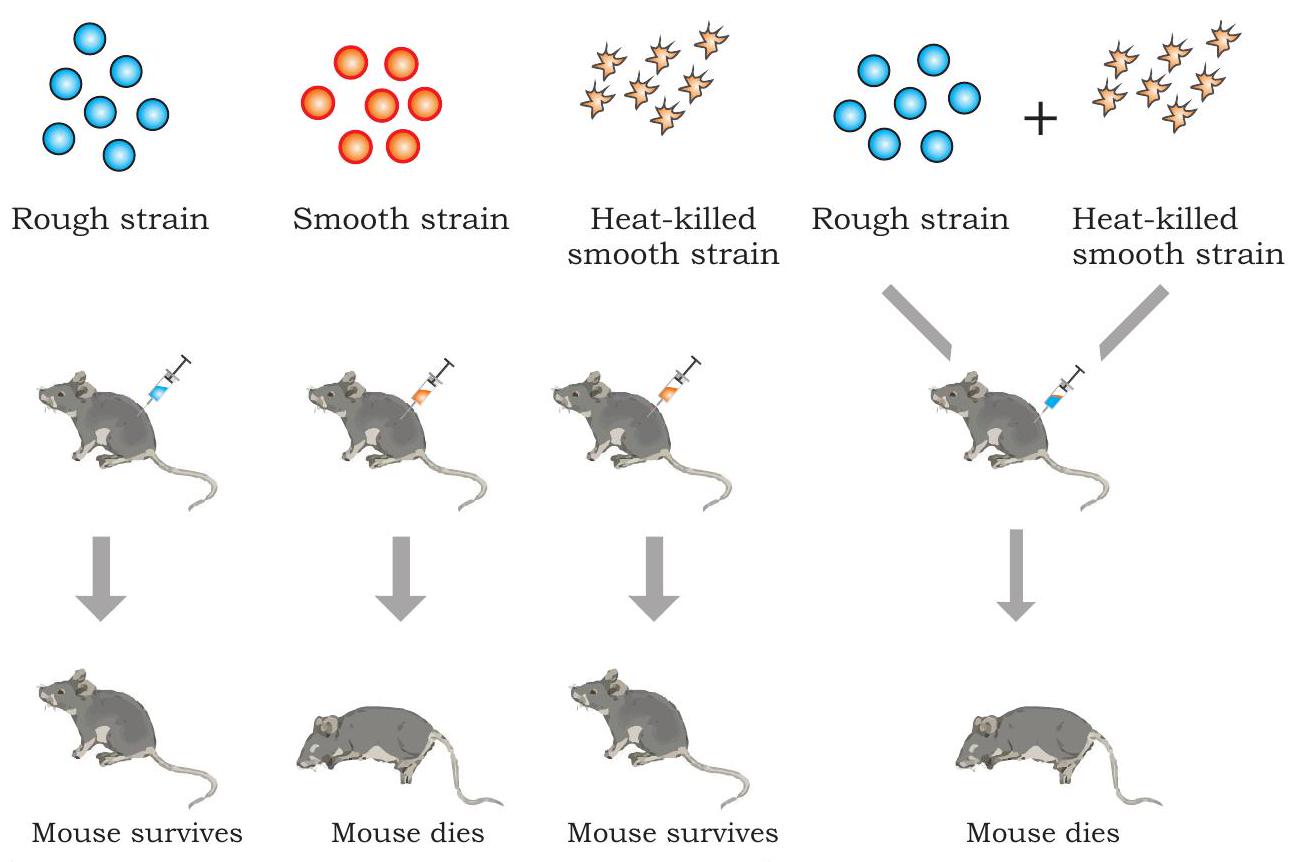
चित्र 7.1: ग्रिफ़िथ का रूपांतरण प्रयोग
आगे समझने के लिए, ग्रिफ़िथ ने कुछ विषाणुकारी $\mathrm{S}$ जीवाणुओं को उबालकर मार दिया और उक्त ताप-हत्या किए गए जीवाणुओं को चूहों में इंजेक्ट किया। उनकी अपेक्षाओं के अनुसार, चूहे बच गए। हालांकि, काफी अप्रत्याशित रूप से, जब ताप-हत्या किए गए $\mathrm{S}$ जीवाणुओं और जीवित $\mathrm{R}$ जीवाणुओं के मिश्रण को इंजेक्ट किया गया, तो चूहे निमोनिया से मर गए। मृत चूहों के रक्त और ऊतक द्रव की जांच से जीवित $\mathrm{S}$ प्रकार के जीवाणुओं की उपस्थिति का पता चला। उपरोक्त प्रेक्षण के आधार पर, ग्रिफ़िथ ने निष्कर्ष निकाला कि R-स्ट्रेन जीवाणुओं ने ताप-हत्या किए गए $S$ जीवाणुओं से उसके द्वारा ‘रूपांतरण सिद्धांत’ कहे गए तत्व को ग्रहण किया होगा, जिससे वे चिकनी कोटेड जीवाणुओं में ‘रूपांतरित’ होकर विषाणुकारी बन गए। उन्होंने इस घटना को रूपांतरण कहा, जिसका अर्थ है एक कोशिका से दूसरी कोशिका में आनुवंशिक पदार्थ का स्थानांतरण जो प्राप्तकर्ता कोशिका की आनुवंशिक संरचना को बदल देता है। लेकिन रूपांतरण करने वाले पदार्थ की प्रकृति अभी भी निर्धारित करनी थी।
7.1.2 रूपांतरण सिद्धांत का जैवरासायनिक वर्णन
तीन वैज्ञानिकों, ओसवाल्ड टी. एवरी, कॉलिन मैकलियोड और मैकलिन मैककार्टी ने ग्रिफ़िथ के रूपांतरण सिद्धांत की पहचान करने के लिए एक श्रृंखला प्रयोग किए, और 1944 में पुष्टि हुई कि रूपांतरण करने वाला कारक DNA है (चित्र 7.2)। प्रयोग की रचना में उन्होंने बैक्टीरिया के स्मूथ स्ट्रेन के तीन मुख्य घटकों, अर्थात् DNA, RNA और प्रोटीन पर ध्यान केंद्रित किया। उन्होंने बैक्टीरिया के हीट-किल्ड स्मूथ स्ट्रेन का एक अर्क तैयार किया जिससे लिपिड और कार्बोहाइड्रेट निकाल दिए गए। अर्क के शेष घटकों में प्रोटीन, RNA और DNA को आगे के प्रयोग के लिए अर्क को तीन भागों में बाँटकर रखा गया। इन अर्कों को अलग-अलग हाइड्रोलिटिक एंजाइमों जैसे राइबोन्यूक्लिएस (RNase), डीऑक्सीराइबोन्यूक्लिएस (DNase) और प्रोटिएस के साथ उपचारित किया गया ताकि क्रमशः RNA, DNA और प्रोटीन को अपघटित किया जा सके, और उनकी रूपांतरण क्षमता का परीक्षण करने के लिए प्रत्येक एंजाइम-उपचारित अर्क को रफ स्ट्रेन के बैक्टीरिया की तीन अलग-अलग संस्कृतियों में स्थानांतरित किया गया। रफ स्ट्रेन का स्मूथ स्ट्रेन में रूपांतरण उन कॉलोनियों में देखा गया जिनमें RNase और प्रोटिएस उपचारित अर्क डाले गए थे, लेकिन उस कॉलोनी में नहीं जिसमें DNase उपचारित अर्क डाला गया था। इन परिणामों ने निस्संदेह स्थापित किया कि DNA ही रूपांतरण सिद्धांत के रूप में कार्य करता है।
परिकल्पना: कोशिकाओं का आनुवंशिक पदार्थ या तो प्रोटीन या न्यूक्लिक अम्ल (DNA या RNA) है

निष्कर्ष: रूपांतरण के लिए DNA आवश्यक है, इसलिए यह कोशिका की आनुवंशिक सामग्री है
चित्र 7.2: रूपांतरित सिद्धांत की पुष्टि
7.1.3 हर्शी-चेस प्रयोग
बाद में, अल्फ्रेड हर्शी और मार्था चेस (1952) द्वारा T2 बैक्टीरियोफेज के साथ किए गए एक और प्रयोग ने DNA को आनुवंशिक सामग्री के रूप में समर्थन देने वाले प्रमाण प्रदान किए। T2 बैक्टीरियोफेज नामक वायरस जो एशेरिखिया कोलाई (Escherichia coli) बैक्टीरिया को संक्रमित करता है, उसमें DNA प्रोटीन कोट से घिरा होता है। जब यह बैक्टीरियल कोशिका को संक्रमित करता है, तो यह बाहरी सतह से जुड़ता है और फिर अपना DNA कोशिका में इंजेक्ट करता है। T2 बैक्टीरियोफेज और E. coli के साथ अपने प्रयोगों की श्रृंखला में उद्देश्य यह स्थापित करना था कि कौन-सा घटक—DNA या प्रोटीन—फेज कणों की वृद्धि के लिए उत्तरदायी है। आसानी से पहचानने के लिए, T2 बैक्टीरियोफेजों को प्रारंभ में E. coli के कालोनियों के साथ रेडियोधानी फॉस्फोरस (³²P) और रेडियोधानी सल्फर (³⁵S) युक्त माध्यम में अलग-अलग उगाया गया (चित्र 7.3)। इससे एक समूह के बैक्टीरियोफेज रेडियोधानी फॉस्फोरस (³²P) से और दूसरे समूह रेडियोधानी सल्फर (³⁵S) से लेबल हो गए।
${ }^{35} \mathrm{S}$ और ${ }^{32} \mathrm{P}$ लेबल किए गए T2 फेजों को अब अलग-अलग दो संस्कृतियों में अनलेबल किए गए E. coli जीवाणु कॉलोनी में इनोक्युलेट किया गया। संक्रमण के बाद, जीवाणु कॉलोनियों को ब्लेंडर में हिलाया गया ताकि जीवाणु कोशिकाओं के बाहर रह गए किसी भी फेज और फेज के अंशों को हटाया जा सके। ब्लेंडर के मिश्रण को फिर सेंट्रीफ्यूज किया गया ताकि जीवाणुओं (जो पेलेट में मौजूद थे) को फेज के मलबे (जो सुपरनेटेंट में मौजूद थे) से अलग किया जा सके। जीवाणु संस्कृति के पेलेट जिनमें रेडियोधर्मिता दिखाई दी, वे रेडियोधर्मी डीएनए वाले फेजों से संक्रमित थे, जबकि ${ }^{35} \mathrm{S}$ बैक्टीरियोफेज से संक्रमित सुपरनेटेंट में रेडियोधर्मिता देखी गई। इससे संकेत मिलता है कि फेज से प्रोटीन जीवाणु में प्रवेश नहीं करते। इसलिए यह निष्कर्ष निकाला गया कि जो पदार्थ जीवाणु कोशिका में प्रवेश करता है, अर्थात् डीएनए, वही आनुवंशिक पदार्थ हो सकता है।
यद्यपि उपरोक्त प्रयोगों ने डीएनए को आनुवंशिक पदार्थ के रूप में प्रबल प्रमाण प्रदान किए, यह स्पष्ट नहीं था कि डीएनए अणु ही आनुवंशिक सूचना का भंडार है। बाद में एरविन चारगाफ, मॉरिस विल्किन्स, रोज़लिंड फ्रैंकलिन, जेम्स वॉटसन और फ्रांसिस क्रिक द्वारा किए गए अध्ययनों ने डीएनए की संरचना की खोज की, जिससे यह स्पष्ट हुआ कि डीएनए बड़ी मात्रा में सूचना को कैसे एन्कोड कर सकता है (जिसका वर्णन अध्याय 3 में किया गया है)।
$\hspace{3.5cm}$बैक्टीरियोफेज

चित्र 7.3: हर्शे-चेस प्रयोग
7.2 प्रोकैरियोटिक और यूकैरियोटिक जीन संगठन
यह स्पष्ट रूप से समझा जाता है कि लक्षण ‘जीन इकाई’ के रूप में माता-पिता से संतानों में प्राप्त होते हैं और डीएनए सभी जीवों में आनुवंशिक पदार्थ है, कुछ वायरसों को छोड़कर (जहाँ आनुवंशिक पदार्थ आरएनए है)। इससे जीन के संगठन के बारे में प्रश्न उठा, क्या यह संगठन प्रोकैरियोट्स में भी यूकैरियोट्स की तरह समान है और यह अणु स्तर पर कैसे कार्य करता है? जीन वंशानुक्रम की वह इकाई है जो एक विशिष्ट लक्षण या विशेषता को नियंत्रित करती है और इसे वैकल्पिक रूपों में भी व्यक्त किया जा सकता है जिन्हें एलील कहा जाता है। दूसरे शब्दों में, जीन डीएनए का एक खंड है जो आरएनए संश्लेषण के माध्यम से पॉलीपेप्टाइड श्रृंखला के संश्लेषण के जरिए स्वयं को व्यक्त करता है, जिसे आण्विक जीव विज्ञान का ‘केन्द्रीय सिद्धांत’ कहा जाता है।
शुरुआत में यह स्थापित किया गया था कि लक्षण या विशेषताओं का विनियमन जीन द्वारा होता है, जो 1900 में मेंडल के वंशागति के सिद्धांत की पुनःखोज के आधार पर था, और इसके बाद पौधों और जानवरों दोनों में हुई एक श्रृंखला अनुसंधानों ने इस तथ्य को स्थापित किया कि लक्षण या विशेषताओं का विनियमन और नियंत्रण कुछ अंतर्निहित सिद्धांत द्वारा होता है जो एक पीढ़ी से दूसरी पीढ़ी तक स्थानांतरित होते हैं। कारक या अंतर्निहित इकाई जो लक्षण या विशेषताओं को नियंत्रित करती है, को बाद में विल्हेल्म जोहानसन ने 1909 में ‘जीन’ नाम दिया।
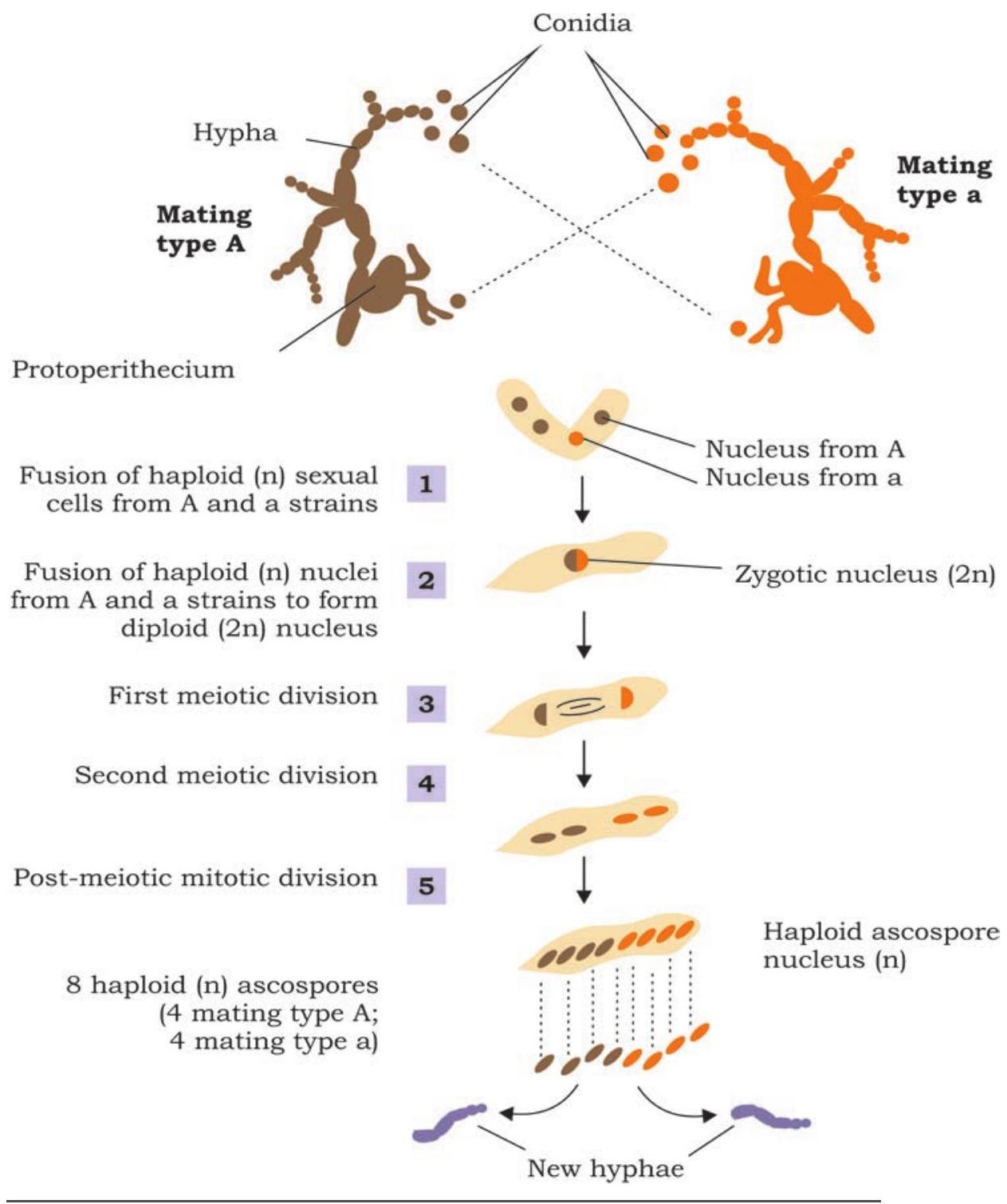
जीन की प्रकृति और कार्यप्रणाली को समझना बीसवीं सदी की शुरुआत के दौरान वैज्ञानिक समुदाय के मुख्य केंद्रों में से एक था। 1930 के दशक में जॉर्ज बीडल और एडवर्ड टैटम ने फफूंद न्यूरोस्पोरा क्रासा पर काम किया, जिसने एक जीन द्वारा एक एंजाइम के संश्लेषण को नियंत्रित करने के संबंध को स्थापित करने में मदद की।
यह विचार करते हुए कि फफूंद Neurospora crassa को सरल शर्करा, अकार्बनिक लवण और विटामिन बायोटिन युक्त माध्यम पर बहुत आसानी से उगाया जा सकता है, बीडल और टैटम ने इस धारणा पर प्रयोग किया कि यह जीव अन्य आवश्यक अमीनो अम्ल और नाइट्रोजनीय क्षारकों का संश्लेषण स्वयं कर सकता है (चित्र 7.4)। यह स्पष्ट माना गया कि संश्लेषण एंजाइमों द्वारा मध्यस्थित होता है, जो जीन नियंत्रण के अंतर्गत संश्लेषित होते हैं। प्रयोग की रचना बहुत सरल थी जिसमे कोनिडिया, अर्थात् अलैंगिक बीजाणुओं को उत्परिवर्तन प्रेरित करने के लिए एक्स-रे से विकिरणित किया गया (चित्र 7.4)। विकिरणित बीजाणुओं द्वारा उत्पन्न संतानों की पहचान उन्हें किसी विशिष्ट न्यूनतम माध्यम पर उगाकर की गई। उत्परिवर्तन युक्त किस्मों की पहचान के उद्देश्य से, विकिरणित बीजाणुओं की संतानों को जंगली प्रकार के साथ संकरित किया गया और उत्तरोत्तर संतानों को न्यूनतम माध्यम पर विशिष्ट अमीनो अम्ल या विटामिन के लिए उगाकर उत्परिवर्ती किस्मों की पहचान की गई (संवर्धन माध्यम जिसमें सभी अमीनो अम्ल, नाइट्रोजनीय क्षारक और विटामिन होते हैं सिवाय किसी विशिष्ट विटामिन या अमीनो अम्ल के)। उन्होंने ऐसे कई उत्परिवर्तनों की पहचान की और जेनेटिक रूप से स्थापित किया कि प्रत्येक उत्परिवर्तन वास्तव में किसी विशिष्ट एंजाइम की अक्रियता का कारण बनता है।
अनुवाद (हिन्दी):
चित्र 7.4: न्यूरोस्पोरा में उत्परिवर्तन के पता लगाने का प्रयोग
इसके बाद यह देखा गया कि सभी प्रोटीन एकल पॉलीपेप्टाइड से नहीं बनते, बल्कि कई प्रोटीन एक से अधिक पॉलीपेप्टाइड श्रृंखलाओं से बने होते हैं। यह तथ्य कि एक जीन एक पॉलीपेप्टाइड को कूटबद्ध करता है; केंद्रीय सिद्धांत (central dogma) को भी “एक जीन-एक प्रोटीन” से संशोधित कर “एक जीन-एक पॉलीपेप्टाइड” कर दिया गया।
लगभग एक ही समय, 1940 के दशक के आरंभ में, क्रोमेटिन तंतु (chromatin fibre) की कोशिकाविज्ञानी जाँचों ने इलेक्ट्रॉन सूक्ष्मदर्शी (electron microscopy) द्वारा “डोरियों पर मनके” जैसी संरचना (चित्र 7.5) प्रकट की और यह तुरंत निष्कर्षित किया गया कि प्रत्येक मनका संभवतः एक जीन को दर्शाता है।
बाद की जांचों से पता चला कि प्रत्येक मनका एक न्यूक्लियोसोम है (जिसमें हिस्टोन अष्टक का एक केंद्र और 146 bp द्विगुणित DNA होता है) और दो मनकों के बीच एक डोरी, लिंकर DNA है। यह भी स्थापित हुआ कि प्रत्येक न्यूक्लियोसोम अपने लिंकर क्षेत्र सहित लगभग 200 bp शामिल करता है। इसे एक जीन नहीं माना जा सकता, क्योंकि कई मामलों में जीन का आकार 200 न्यूक्लियोटाइड से कहीं अधिक बड़ा होना चाहिए। सरल कारण यह है कि कई प्रोटीनों में 100 से अधिक अमीनो अम्ल अवशेष होते हैं और उनके संगत नियामक जीन उससे तीन गुना कम नहीं हो सकते (कोडॉन की त्रिक प्रकृति के आधार पर)।
यह अब स्पष्ट हो गया है कि एक जीन DNA के उस खंड होता है जिसमें एक विशिष्ट प्रमोटर क्षेत्र होता है, जहाँ RNA पॉलिमरेज बाइंड कर सकता है और mRNA का ट्रांसक्रिप्शन कर सकता है। ट्रांसक्राइब किया गया mRNA फिर अनुवाद की प्रक्रिया में शामिल होता है। यह तंत्र वायरस से लेकर बैक्टीरिया, पौधों और जानवरों तक सभी जीवों के लिए समान है। क्या किसी वायरस, बैक्टीरिया या उच्च जीव के जीन उनकी संरचना और कार्य में समान होते हैं? एक पूर्ण सेट गुणसूत्रों में मौजूद कुल DNA सामग्री (सभी जीनों और DNA के अन्य भागों सहित) को जीनोम कहा जाता है। जैसा कि हम वायरस या बैक्टीरिया के मामले में देखते हैं, जीनोम का आकार यूकैरियोटिक जीनोम की तुलना में अपेक्षाकृत बहुत छोटा होता है। यूकैरियोटिक जीनोम प्रोकैरियोटिक जीनोम की तुलना में बहुत अधिक जटिल होते हैं। पौधों के जीनोम अन्य किसी भी यूकैरियोटिक जीनोम की तुलना में और भी अधिक जटिल होते हैं। विभिन्न समूहों के जीवित जीवों में जीनोम के अनुमानित आकार को चित्र 7.6 में दिखाया गया है।
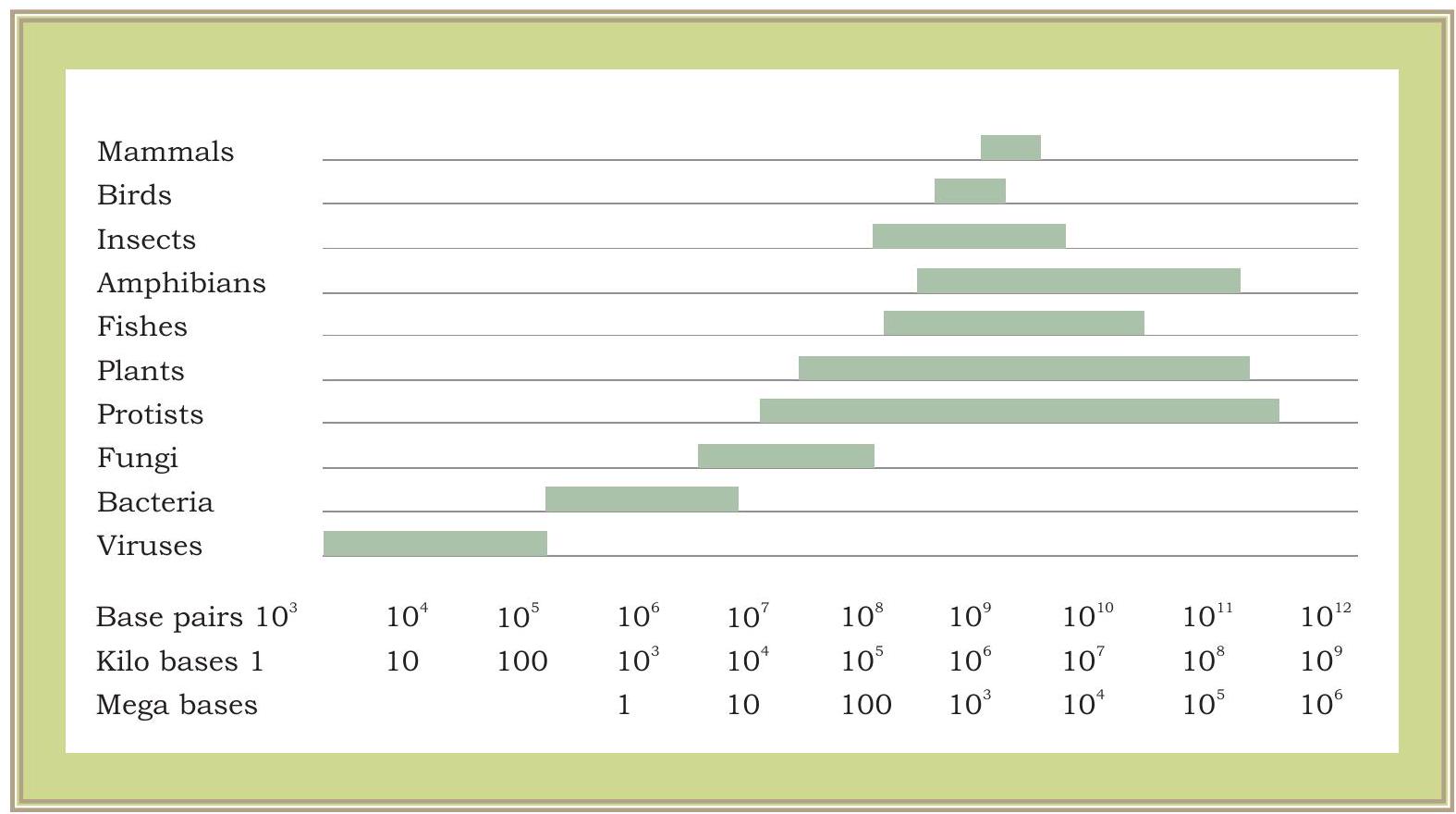
चित्र 7.6: विभिन्न समूहों के जीवित जीवों में जीनोम आकार में भिन्नता। जीनोम आकारों को न्यूक्लियोटाइड युग्मों के हजारों में मापा जाता है, अर्थात् $1000 \mathrm{bp}=1$ किलोबेस (kb) और 1000,000 $\mathrm{bp}=1000 \mathrm{~Kb}=1$ मेगाबेस (Mb)
अधिकांश यूकैरियोट्स में जीनोम का एक बड़ा भाग अभिव्यक्त नहीं होता और गैर-कोडिंग अनुक्रम के रूप में बना रहता है। यह भी देखा गया है कि यूकैरियोटिक जीन अभिव्यक्ति में पॉलीपेप्टाइड श्रृंखला संश्लेषण की प्रक्रिया में शामिल सक्रिय mRNA का आकार प्राथमिक ट्रांसक्रिप्ट से बहुत छोटा होता है। वास्तव में, कई यूकैरियोटिक जीन, उदाहरण के लिए, हीमोग्लोबिन का $\beta$-ग्लोबिन जीन, ट्रांसक्रिप्शन के बाद स्प्लाइसिंग की प्रक्रिया से गुजरता है जिसमें प्राथमिक ट्रांसक्रिप्ट के कुछ बीच-बीच वाले खंड (इंट्रॉन) को काटकर निकाल दिया जाता है और शेष भाग (एक्सॉन) को आपस में जोड़कर mRNA बनाया जाता है।
एक यूकैरियोटिक कोल में दो प्रकार के जीनोम होते हैं: (i) केंद्रक जीनोम और (ii) अंगक जीनोम।
केंद्रक जीनोम
DNA का अधिकांश भाग केंद्रक में पाया जाता है और इसे केंद्रक DNA कहा जाता है। प्रोकैरियोट्स में, अधिकांश जीनोम कोडिंग DNA अनुक्रमों से बना होता है जबकि यूकैरियोटिक जीनोम में कोडिंग क्षेत्र कुल जीनोम का अपेक्षाकृत बहुत छोटा भाग बनाते हैं। उदाहरण के लिए, मानव जीनोम का आकार लगभग $3,000 \mathrm{Mb}$ या 3 अरब DNA बेस युग्म है और अनुमानतः इसमें 20,000 से अधिक जीन हैं, जो कुल जीनोम का लगभग $2 %$ बनाते हैं। जीनोम के गैर-कोडिंग क्षेत्र में ऐसे अनुक्रम होते हैं जो हजारों से लेकर कई लाख बार टैंडम ऐरे के रूप में दोहराए जाते हैं। इन दोहराए जाने वाले DNA अनुक्रमों का आकार और संख्या जीनोम में काफी भिन्न होती है।
यूकैरियोटिक कोशिका में नाभिकीय जीनोम छोटे संघनित इकाइयों में संगठित होता है, जिन्हें गुणसूत्र कहा जाता है जिनमें रैखिक डीएनए अणु होते हैं। किसी व्यक्ति के हेप्लॉयड जीनोम में गुणसूत्रों का एक पूर्ण समुच्चय अक्षर n द्वारा दर्शाया जाता है। अधिकांश जीव प्रत्येक कोशिका में गुणसूत्रों के दो समुच्चय रखते हैं और इन्हें डिप्लॉयड जीव (2n) कहा जाता है।
अंगाकार जीनोम
नाभिकीय डीएनए के अतिरिक्त, कुछ झिल्लीबद्ध कोशिकीय अंगिकाएँ जैसे क्लोरोप्लास्ट और माइटोकॉन्ड्रिया अंग-विशिष्ट डीएनए रखती हैं। अंग जीनोम अधिकांशतः वृत्ताकार द्वि-स्तंभी डीएनए होते हैं और प्रत्येक अंग में एकाधिक प्रतियों में मौजूद होते हैं (चित्र 7.7)। ये अर्ध-संरक्षित तरीके से प्रतिकृत होते हैं और नाभिकीय जीनोम से पृथक रूप से वंशानुगत होते हैं। अंग डीएनए में ऐसे जीन होते हैं जो अंग-विशिष्ट कार्यों के लिए आवश्यक होते हैं और ये सामान्यतः एकल-पैतृक होते हैं तथा अगली पीढ़ी में मादा युग्मकों के माध्यम से वंशानुगत होते हैं।

चित्र 7.7: अंगाकार डीएनए
7.3 डीएनए प्रतिकृति
जब जेम्स वॉटसन और फ्रांसिस क्रिक ने 1953 में डीएनए की त्रि-आयामी संरचना प्रस्तावित की, तो जीवविज्ञानियों को सबसे अधिक उत्साहित करने वाली विशेषता दो पॉलीन्यूक्लियोटाइड श्रृंखलाओं के आधारों की पूरक संबंध थी। वॉटसन और क्रिक ने तुरंत एक मॉडल सुझाया कि आनुवंशिक सूचना की प्रतिलिपि बनाने का आधार पूरकता है (विवरण के लिए इकाई II, अध्याय 2 देखें)। उनके द्वारा प्रस्तावित मॉडल के अनुसार, डीएनए की दोनों स्ट्रैंड प्रतिकृतिकरण के दौरान अलग हो जाती हैं; प्रत्येक स्ट्रैंड एक नई पूरक स्ट्रैंड के संश्लेषण के लिए टेम्पलेट का कार्य करती है क्योंकि आधार युग्मन की विशिष्टता होती है (अर्थात् थाइमिन एडेनिन के साथ और साइटोसिन ग्वानिन के साथ)। इस प्रकार माता-पिता के द्विसंकरी डीएनए से दो समान पुत्री द्विसंकरी बनती हैं, जिनमें से प्रत्येक में एक माता-पिता की स्ट्रैंड और एक नव-संश्लेषित पुत्री स्ट्रैंड होती है। डीएनए प्रतिकृतिकरण का यह रूप अर्ध-संरक्षी प्रतिकृतिकरण कहलाता है (चित्र 7.8)। इस प्रतिकृतिकरण विधि के प्रमाण मैथ्यू मेसेलसन और फ्रैंकलिन स्टाहल ने 1958 में प्रदान किए।
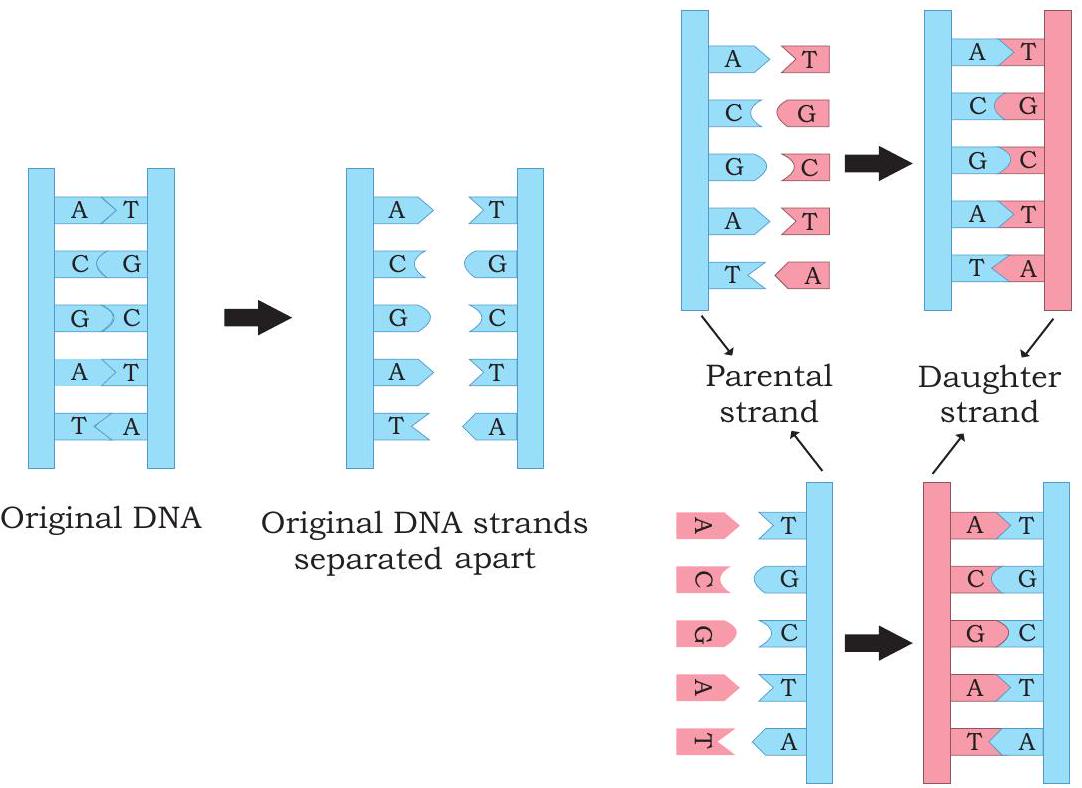
चित्र 7.8: डीएनए प्रतिकृतिकरण का अर्ध-संरक्षी प्रकार
7.3.1 मेसेलसन और स्टाहल प्रयोग
पुराने और नए स्ट्रैंड के बीच अंतर करने के लिए, मेसेल्सन और स्टाहल ने नाइट्रोजन के दो समस्थानिकों का उपयोग किया, ${ }^{14} \mathrm{~N}$ (सामान्य रूप) और ${ }^{15} \mathrm{~N}$ (दुर्लभ, भारी रूप)। उन्होंने $E$. कोलाई बैक्टीरिया को नाइट्रोजन के भारी समस्थानिक को एकमात्र नाइट्रोजन स्रोत के रूप में युक्त माध्यम में उगाया। कई पीढ़ियों के बाद, सभी $E$. कोलाई कोशिकाओं में ${ }^{15} \mathrm{~N}$ डीएनए के प्यूरीन और पिरिमिडीन बेस में सम्मिलित हो गया। घनत्व ग्रेडिएन्ट सेंट्रिफ्यूगेशन का उपयोग करके, उन्होंने देखा कि ${ }^{15} \mathrm{~N}$ में उगाए गए $E$. कोलाई से निकाले गए डीएनए ने सेंट्रिफ्यूज ट्यूब के निचले हिस्से में एकल बैंड उत्पन्न किया, जबकि ${ }^{14} \mathrm{~N}$ माध्यम में उगाई गई बैक्टीरियल कोशिकाओं से निकाले गए डीएनए ने ऊपर की ओर एक बैंड बनाया। इससे संकेत मिला कि ${ }^{15} \mathrm{~N}$ माध्यम में उगाई गई $E$. कोलाई कोशिकाओं का डीएनए हल्के समस्थानिक $\left({ }^{14} \mathrm{~N}\right)$ युक्त माध्यम में उगाए गए बैक्टीरिया के डीएनए से अधिक घना था (चित्र 7.9)।
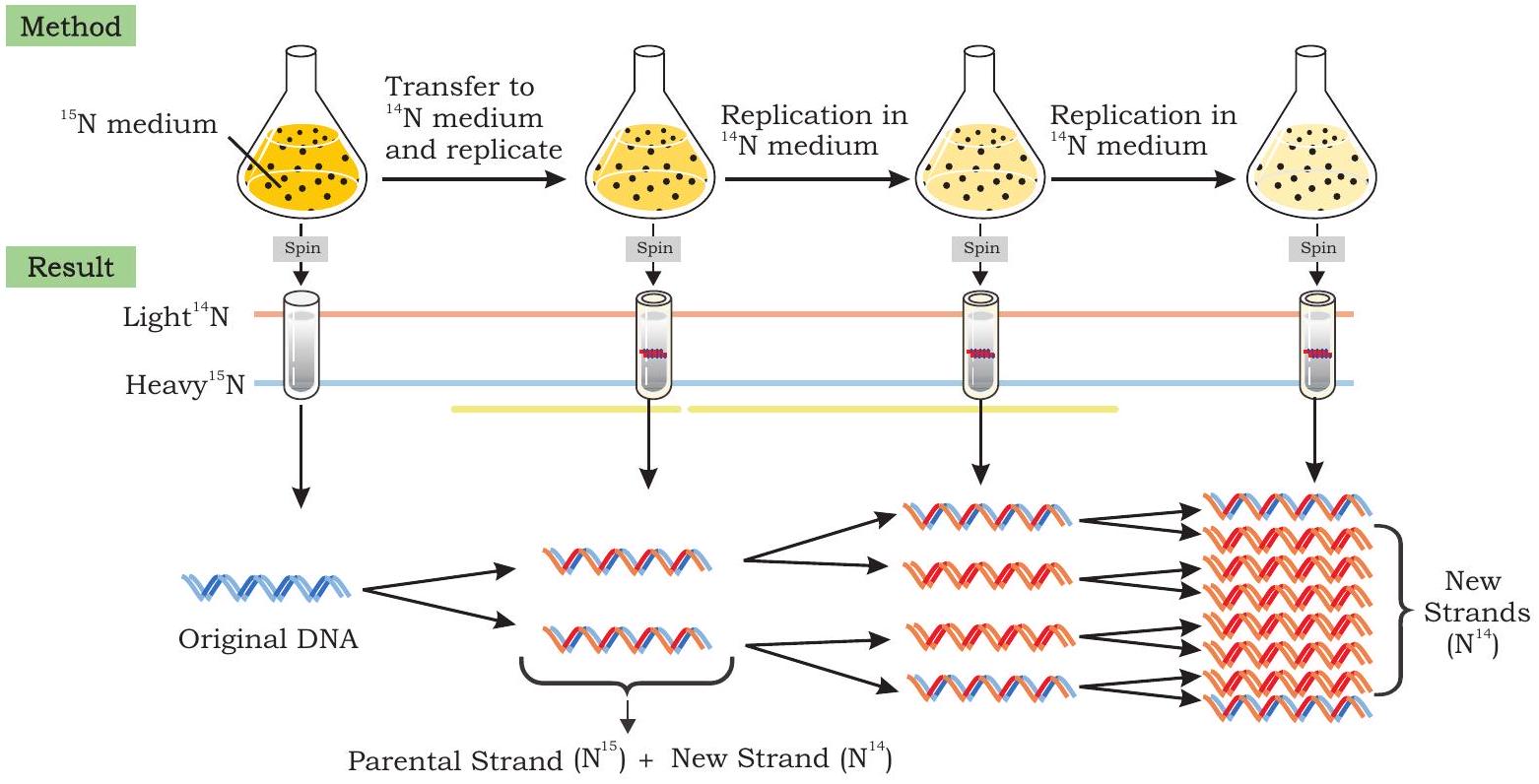
चित्र 7.9: डीएनए प्रतिकृतिकरण के अर्ध-संरक्षित मोड की पुष्टि करने के लिए मेसेल्सन-स्टाहल प्रयोग
मेसेल्सन और स्टाहल ने तब E. coli बैक्टीरिया को ${ }^{15} \mathrm{~N}$ माध्यम से ${ }^{14} \mathrm{~N}$ माध्यम में स्थानांतरित किया और बैक्टीरियल कोशिकाओं के गुणन के दौरान विभिन्न समय अंतरालों पर DNA एकत्र किया (E. coli हर 20 मिनट में विभाजित होता है)। पहले विभाजन चक्र (पीढ़ी I) के बाद E. coli कोशिकाओं से निकाले गए DNA का विश्लेषण सीज़ियम क्लोराइड लवण का उपयोग कर घनत्व ग्रेडिएन्ट सेंट्रीफ्यूगेशन द्वारा किया गया तो एकल बैंड उत्पन्न हुआ, लेकिन भारी DNA $\left({ }^{15} \mathrm{~N}\right)$ और हल्के DNA $\left({ }^{14} \mathrm{~N}\right)$ बैंडों के बीच की स्थिति पर।

मैथ्यू मेसेल्सन और फ्रैंकलिन स्टाहल ने DNA प्रतिकृतिकरण के अर्ध-संरक्षित प्रकार की पुष्टि की
जब DNA को ${ }^{14} \mathrm{~N}$ माध्यम में दूसरे प्रतिकृतिकरण चक्र (पीढ़ी II) के बाद E. coli कोशिकाओं से निकाला गया, तो सेंट्रीफ्यूज ट्यूब में दो समान तीव्रता के बैंड दिखाई दिए, एक मध्यम स्थिति पर और दूसरा उस स्थिति पर जो केवल ${ }^{14} \mathrm{~N}$ DNA वाले DNA की अपेक्षित होती है। जब DNA के नमूने अतिरिक्त प्रतिकृतिकरण चक्रों के बाद एकत्र किए गए, तो उन्होंने दो बैंड उत्पन्न किए। हल्के DNA का प्रतिनिधित्व करने वाले बैंड धीरे-धीरे मोटे होते गए, लेकिन मध्यम स्थिति पर बैंड अपरिवर्तित रहा।
बॉक्स 1
घनत्व प्रवणी केन्द्रापसारण (Density Gradient Centrifugation)
यह एक केन्द्रापसारण तकनीक है जिसका उपयोग मिश्रण में उपस्थित अणुओं को उनके घनत्व के आधार पर पृथक करने के लिए किया जाता है। केन्द्रापसारण नलिका को सीज़ियम क्लोराइड ( $\mathrm{CsCl}$ ) के भारी लवण विलयन और जिस नमूने का घनत्व मापना है, उससे भरा जाता है। फिर नलिका को एक अल्ट्रासेंट्रीफ्यूज में कई दिनों तक बहुत उच्च चाल से घुमाया जाता है। अल्ट्रासेंट्रीफ्यूज द्वारा उत्पन्न विशाल कृत्रिक बल Cs आयनों को नलिका के तल की ओर प्रवास करने को बाध्य करता है, जिससे तल पर उच्च घनत्व और ऊपर कम घनत्व वाला एक प्रवणी (gradient) बनता है। DNA स्ट्रैंड इस प्रवणी में तैरते या डूबते हैं जब तक कि उनका घनत्व लवण के घनत्व से मेल नहीं खाता।
7.3.2 मेसेल्सन और स्टाहल द्वारा परिणामों की व्याख्या
प्रतिकृतिकरण (replication) के पहले चक्र के बाद, प्रत्येक पुत्री DNA द्विगुणक (duplex) एक संकर (hybrid) थी जिसमें एक भारी स्ट्रैंड जो मूल से ${ }^{15} \mathrm{~N}$ युक्त था और एक हल्का स्ट्रैंड जो माध्यम से ${ }^{14} \mathrm{~N}$ युक्त था। जब यह संकर द्विगुणक पुनः प्रतिकृत हुआ, तो भारी टेम्पलेट स्ट्रैंड ने एक और संकर द्विगुणक DNA बनाया जबकि हल्के स्ट्रैंड ने हल्का DNA द्विगुणक बनाया। इस प्रकार, मेसेल्सन और स्टाहल के प्रयोग ने वाटसन और क्रिक मॉडल की भविष्यवाणी को स्पष्ट रूप से पुष्ट की कि DNA अर्ध-संरक्षित (semiconservative) तरीके से प्रतिकृत होता है।
टेलर और सहयोगियों ने 1958 में भी विकिया फाबा में समान प्रयोग किए, जिन्होंने नवसंश्लेषित डीएनए के वितरण का पता लगाने के लिए रेडियोधर्मी थाइमिडिन का उपयोग किया। उन्होंने यह भी सिद्ध किया कि गुणसूत्रों में डीएनए अर्धसंरक्षित रूप से प्रतिकृत होता है।
7.3.3 प्रतिकृतिकरण की यंत्रिका
द्विसूत्री डीएनए अणु की प्रतिकृति करने के लिए यह अनावृत होना चाहिए ताकि सूत्रों को अलग कर आधारों को उजागर किया जा सके। दोनों डीएनए सूत्र पूरक आधारों की विधान के लिए टेम्पलेट का कार्य करते हैं ताकि नई पॉलीन्यूक्लियोटाइड सूत्र बन सकें जो टेम्पलेट सूत्र के प्रति प्रतिलोम समानांतर होते हैं। डबल हेलिक्स को खोलने और डीएनए की नई सूत्रों के संश्लेषण के लिए कई प्रोटीन और एंजाइमों की आवश्यकता होती है। डीएनए की प्रतिकृति में शामिल प्रमुख एंजाइम और प्रोटीन, जो प्रोकैरियोट्स और यूकैरियोट्स दोनों में होते हैं, इस प्रकार हैं-
(i) डीएनए पॉलिमरेज़
ये प्रतिकृतिकरण के मुख्य एंजाइम हैं क्योंकि ये नए डीएनए सूत्रों का संश्लेषण करने के लिए उत्तरदायी होते हैं। ये $5^{\prime} \rightarrow 3^{\prime}$ दिशा में नए डीएनए सूत्रों का संश्लेषण करते हैं, डीएनए श्रृंखला के बढ़ते सिरे पर स्थित $3^{\prime}$ हाइड्रॉक्सिल समूह और आने वाले डीऑक्सी-राइबोन्यूक्लियोसाइड ट्राइफॉस्फेट (dNTP) के $5^{\prime}$ फॉस्फेट समूह के बीच फॉस्फोडाइएस्टर बंध के निर्माण को उत्प्रेरित करके।
प्रोकैरियोट्स में तीन प्रकार की डीएनए पॉलिमरेज़ होती हैं—डीएनए पॉलिमरेज़ I, II, III। डीएनए पॉलिमरेज़ III डीएनए प्रतिकृतिकरण का मुख्य एंजाइम है। यह नई स्ट्रैंड का संश्लेषण $5^{\prime} \rightarrow 3^{\prime}$ दिशा में करता है और अपनी $3^{\prime} \rightarrow 5^{\prime}$ एक्सोन्यूक्लिएस गतिविधि द्वारा बढ़ती श्रृंखला में त्रुटियों को सुधारता है, गलत तरीके से शामिल न्यूक्लियोटाइड्स को $3^{\prime} \rightarrow 5^{\prime}$ दिशा में हटाकर। डीएनए पॉलिमरेज़ I में $5^{\prime} \rightarrow 3^{\prime}$ पॉलिमराइज़ेशन गतिविधि के अतिरिक्त $5^{\prime} \rightarrow 3^{\prime}$ और $3^{\prime} \rightarrow 5^{\prime}$ दोनों एक्सोन्यूक्लिएस गतिविधियाँ होती हैं। अपनी $5^{\prime} \rightarrow 3^{\prime}$ एक्सोन्यूक्लिएस गतिविधि द्वारा यह प्राइमेज़ द्वारा बनाए गए आरएनए प्राइमरों को हटाता है। डीएनए पॉलिमरेज़ II डीएनए की मरम्मत में संलग्न होता है। यूकैरियोट्स में नई स्ट्रैंड्स के संश्लेषण और क्षतिग्रस्त डीएनए की मरम्मत में कई डीएनए पॉलिमरेज़ शामिल होते हैं।
(ii) प्राइमेज़
डीएनए पॉलिमरेज़ की कमी यह है कि यह डीएनए की नई स्ट्रैंड का प्रारंभ नहीं कर सकता। इसे एक आरएनए प्राइमर चाहिए होता है, न्यूक्लियोटाइड्स का एक मौजूदा खंड जो $3^{\prime} \mathrm{OH}$ समूह प्रदान करता है जिस पर यह एक नया न्यूक्लियोटाइड जोड़ सकता है। प्राइमेज़, एक डीएनए-निर्भर आरएनए पॉलिमरेज़, डीएनए प्रतिकृतिकरण को प्रारंभ करने के लिए लगभग 10-12 न्यूक्लियोटाइड्स लंबी छोटी आरएनए ओलिगोन्यूक्लियोटाइड श्रृंखला, जिसे आरएनए प्राइमर कहा जाता है, का संश्लेषण करता है।
(iii) हेलिकेसेज़
एकल स्ट्रैंड टेम्प्लेट बनाने के लिए, डीएनए डबल हेलिक्स को खोलना पड़ता है। डीएनए हेलिकेस, एटीपी के रूप में ऊर्जा का उपयोग करके, डीएनए अणु की दोनों स्ट्रैंड्स के बीच मौजूद हाइड्रोजन बॉन्ड्स को तोड़ता है।
(iv) टोपोइसोमरेज
जैसे-जैसे डीएनए की दोनों स्ट्रैंड्स अलग होती हैं, टॉर्शनल स्ट्रेन के कारण डीएनए हेलिक्स कॉइल होकर रेप्लिकेशन फोर्क के सामने गांठ बना लेती है। टोपोइसोमरेज़ इस टेंशन को दूर करते हैं हेलिक्स को उसकी कॉइल्ड और सुपरकॉइल्ड संरचना में रखने वाले डीएनए को निक करके और फिर से जोड़कर।
(v) सिंगल स्ट्रैंड बाइंडिंग (SSB) प्रोटीन
हेलिकेस द्वारा डीएनए के अनवाइंडिंग के बाद, एकल स्ट्रैंड न्यूक्लियोटाइड चेनें फिर से हाइड्रोजन बॉन्ड बनाकर री-ऐनील होने की प्रवृत्ति रखती हैं। SSB प्रोटीन उजागर हुए एकल स्ट्रैंड डीएनए से कसकर जुड़ जाते हैं और उन्हें रेप्लिकेशन हो सके इसलिए एकल स्ट्रैंड रूप में स्थिर रखते हैं।
(vi) डीएनए लाइगेज
डीएनए लाइगेज एंजाइम नव-संश्लेषित डीएनए खंडों को $3^{\prime} \mathrm{OH}$ समूह और 5’ फॉस्फेट समूह के बीच फॉस्फोडाइएस्टर बॉन्ड बनाकर जोड़ता है।
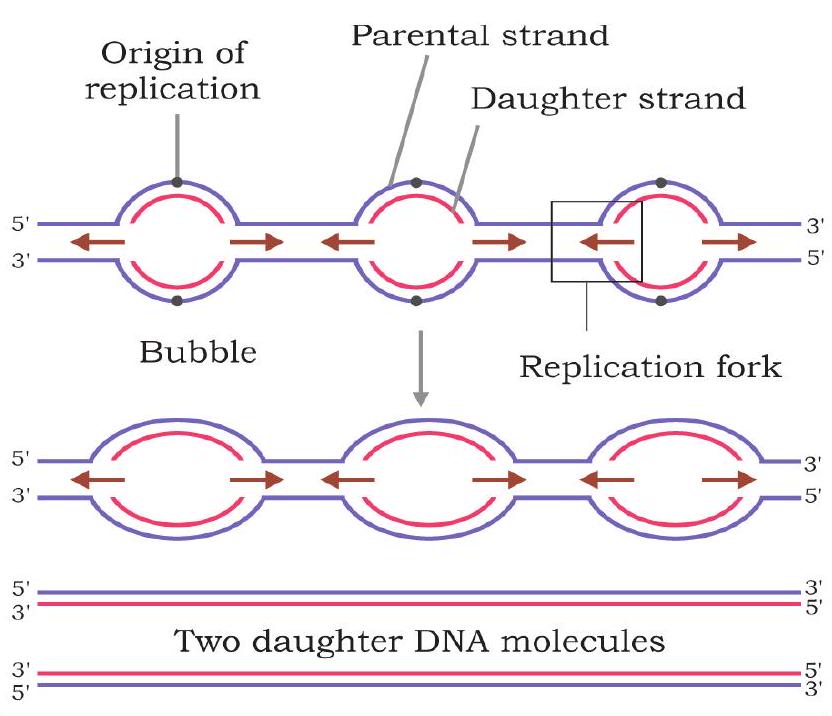
7.3.4 बैक्टीरिया में डीएनए रेप्लिकेशन की क्रियाविधि
रेप्लिकेशन आमतौर पर डीएनए अनुक्रम पर एक विशिष्ट स्थान से शुरू होता है जिसे ऑरिजिन ऑफ रेप्लिकेशन (ori) कहा जाता है (चित्र 7.10)।

चित्र 7.10: बैक्टीरियल क्रोमोसोम्स में रेप्लिकेशन का एक ही प्रारंभिक बिंदु होता है
बैक्टीरिया में, DNA वृत्ताकार और द्वि-स्ट्रैंडेड होता है। इसमें एकल प्रतिकृत्ति उत्पत्ति बिंदु होता है (E. coli में इसे oriC कहा जाता है) और यह एक विशिष्ट स्थल, टर्मिनस (terminus) पर समाप्त होता है। आरंभक प्रोटीन प्रतिकृत्ति के उत्पत्ति बिंदु से बंधते हैं और DNA के एक छोटे से भाग को अनावृत्त करते हैं। आगे, प्रारंभिक उद्घाटन के दोनों ओर दोनों स्ट्रैंडों का पृथक्करण हेलिकेस (helicase) द्वारा किया जाता है, जो दो न्यूक्लियोटाइड स्ट्रैंडों के बीच उपस्थित हाइड्रोजन बंधों को तोड़ता है। दोनों ओर प्रतिकृत्ति कांटे (replication forks) बनते हैं और वे उत्पत्ति बिंदु से विपरीत दिशा में दूर चले जाते हैं। इस प्रतिकृत्ति की विधि को द्विदिशात्मक प्रतिकृत्ति (bidirectional replication) कहा जाता है (चित्र 7.10)। चूंकि DNA संश्लेषण के लिए एकल स्ट्रैंड टेम्प्लेट की आवश्यकता होती है, एकल स्ट्रैंड बंधन प्रोटीन एकल स्ट्रैंड DNA से बंधकर उसे स्थिर करते हैं। एक टोपोइसोमरेज (topoisomerase), DNA स्ट्रैंडों के अनावृत्त होने के परिणामस्वरूप प्रतिकृत्ति कांटे के सामने उत्पन्न होने वाले टॉर्शनल तनाव को कम करता है (चित्र 7.11)।
DNA पॉलिमरेज III एक नया DNA स्ट्रैंड $5^{\prime} \rightarrow 3^{\prime}$ दिशा में, टेम्प्लेट स्ट्रैंड के प्रतिपरallel संश्लेषित करता है (चित्र 7.12)। DNA पॉलिमरेज नग्न टेम्प्लेट पर DNA संश्लेषण प्रारंभ नहीं कर सकता; बल्कि यह प्राइमर स्ट्रैंड के $3^{\prime} \mathrm{OH}$ समूह पर न्यूक्लियोटाइड जोड़ सकता है।

आकृति 7.11: डीएनए संश्लेषण दोनों डीएनए टेम्पलेट स्ट्रैंडों पर एक साथ लेकिन विपरीत दिशाओं में होता है
प्राइमेज एक आरएनए प्राइमर का संश्लेषण करता है, जो मूल बिंदु पर प्रत्येक प्रतिकृतिकां कांटे में टेम्पलेट स्ट्रैंड पर लगभग 10-12 न्यूक्लियोटाइड लंबे $5^{\prime} \rightarrow 3^{\prime}$ दिशा में एक छोटे हिस्से के रूप में होता है। आरएनए प्राइमर 3’ $\mathrm{OH}$ समूह प्रदान करता है जिससे डीएनए पॉलिमरेज न्यूक्लियोटाइड जोड़ता है। प्रतिकृतिकां कांटे में $5^{\prime} \rightarrow 3^{\prime}$ दिशा में उन्मुख टेम्पलेट स्ट्रैंड पर आरएनए प्राइमर के निर्माण के बाद, डीएनए पॉलिमरेज III पॉलिन्यूक्लियोटाइड स्ट्रैंड को लंबा करता है जिससे डीएनए स्ट्रैंड के बढ़ते हुए सिरे पर मौजूद $3^{\prime} \mathrm{OH}$ समूह और आने वाले डिऑक्सीराइबोन्यूक्लियोसाइड ट्राइफॉस्फेट (dNTP) के 5’ फॉस्फेट समूह के बीच फॉस्फोडाइएस्टर बंध के निर्माण की क्रिया को उत्प्रेरित करता है।
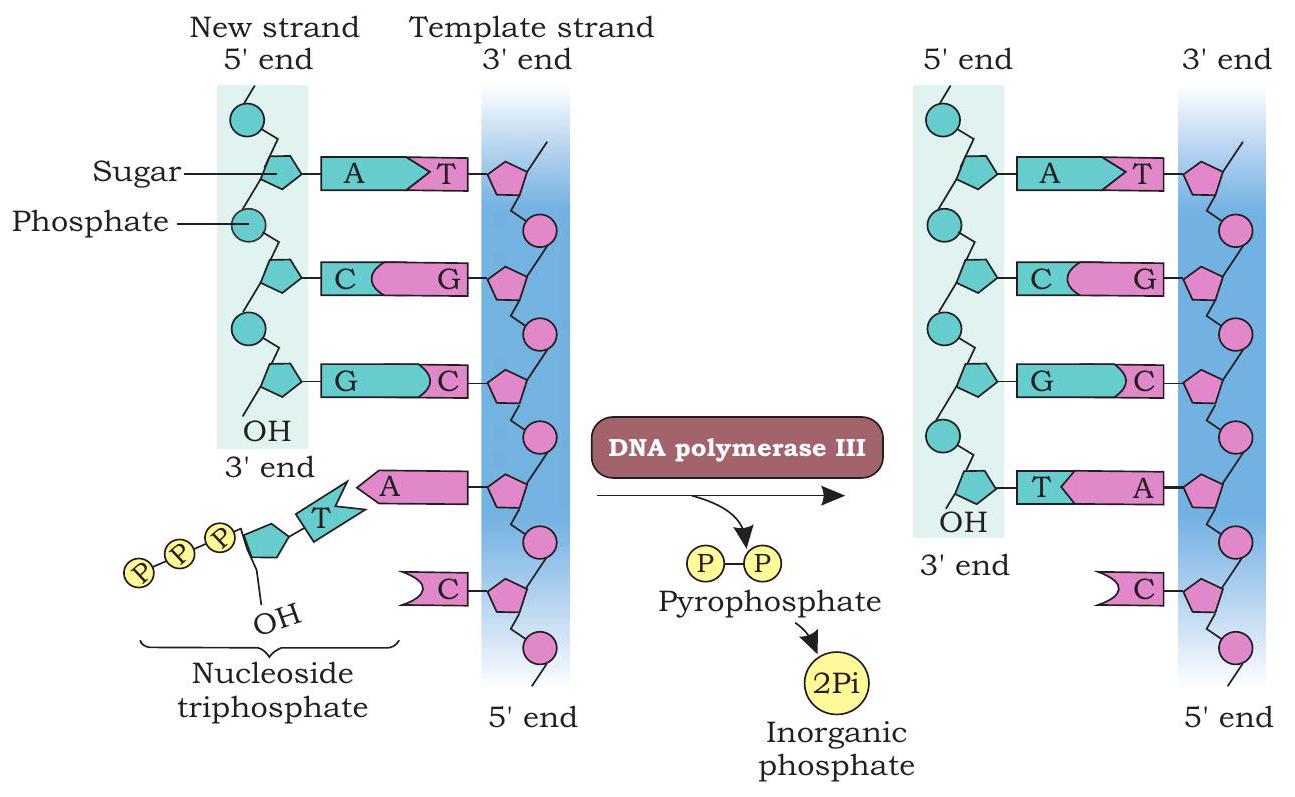
आकृति 7.12: डीएनए पॉलिमरेज पिछले न्यूक्लियोटाइड के 3’ $\mathrm{OH}$ समूह में 5’ फॉस्फेट समूह की जोड़ी की क्रिया को उत्प्रेरित करता है
प्रत्येक चरण में, आने वाले dNTP के $\beta$ और $\gamma$ फॉस्फेट क्लीव किए जाते हैं और परिणामी न्यूक्लियोटाइड बढ़ते हुए न्यूक्लियोटाइड स्ट्रैंड के 3’-OH समूह में जोड़ा जाता है। टेम्पलेट स्ट्रैंड पर, जो $3^{\prime} \rightarrow 5^{\prime}$ दिशा में उन्मुख है, नया स्ट्रैंड $5^{\prime} \rightarrow 3^{\prime}$ दिशा में लगातार संश्लेषित होता है और इसे लीडिंग स्ट्रैंड कहा जाता है (चित्र 7.13)।
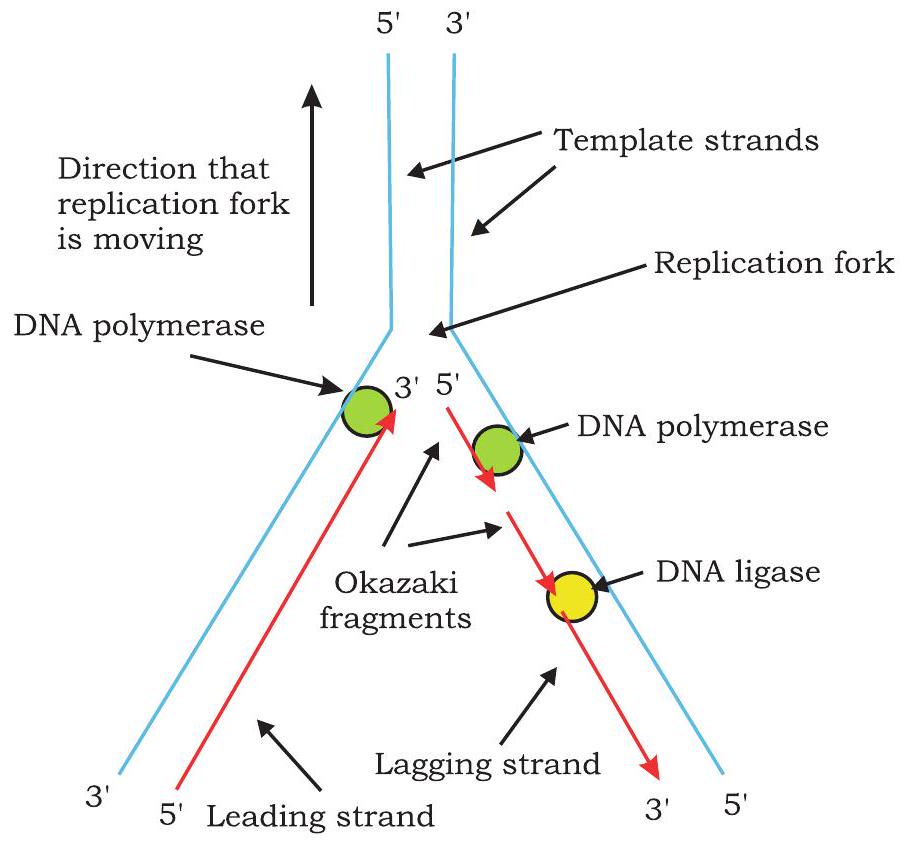
चित्र 7.13: लीडिंग स्ट्रैंड लगातार संश्लेषित होता है और लैगिंग स्ट्रैंड असतत रूप से संश्लेषित होता है
प्रतिकृति कांटे (replication fork) में दोनों टेम्पलेट स्ट्रैंड्स पर नए डीएनए स्ट्रैंड्स का संश्लेषण एक साथ होता है। टेम्पलेट स्ट्रैंड, जो प्रतिकृति कांटे में $5^{\prime} \rightarrow 3^{\prime}$ दिशा में उन्मुख है, पर प्रतिकृति प्रतिकृति कांटे की गति की विपरीत दिशा में आगे बढ़ती है। प्राइमेज (primase) इस टेम्पलेट स्ट्रैंड पर प्रतिकृति कांटे पर एक आरएनए प्राइमर संश्लेषित करता है। डीएनए पॉलिमरेज़ III प्राइमर के $3^{\prime} \mathrm{OH}$ सिरे पर न्यूक्लियोटाइड जोड़ता है और लगभग 1000-2000 न्यूक्लियोटाइड्स का एक छोटा डीएनए खंड संश्लेषित करता है। जैसे-जैसे अनवाइंडिंग आगे बढ़ती है, एक और आरएनए प्राइमर बनता है जिसे डीएनए पॉलिमरेज़ द्वारा बढ़ाया जाता है। इस टेम्पलेट पर संश्लेषण असतत रूप से होता है, छोटे खंडों के रूप में जिन्हें ओकाज़ाकी खंड (Okazaki fragment) कहा जाता है, जिन्हें पहली बार आर. ओकाज़ाकी और सहयोगियों ने पहचाना था। प्रत्येक ओकाज़ाकी खंड एक आरएनए प्राइमर से शुरू होता है। जो डीएनए स्ट्रैंड असतत रूप से संश्लेषित होता है उसे लैगिंग स्ट्रैंड (lagging strand) कहा जाता है।
चूँकि डीएनए प्रतिकृतिकरण के दौरान एक सतत रूप से संश्लेषित होती है (लीडिंग स्ट्रैंड) और दूसरी असतत रूप से संश्लेषित होती है (लैगिंग स्ट्रैंड), प्रतिकृतिकरण को अर्ध-असतय कहा जाता है। इस प्रकार, लीडिंग स्ट्रैंड के 5’ सिरे पर केवल एक आरएनए प्राइमर होता है जबकि लैगिंग स्ट्रैंड पर कई आरएनए प्राइमर होते हैं (ओकाज़ाकी खंडों की संख्या के बराबर)। यहाँ प्रश्न उठता है कि इन ओकाज़ाकी खंडों को एक सतत डीएनए स्ट्रैंड में कैसे परिवर्तित किया जाता है? डीएनए पॉलिमरेज़ I अपनी 5’ → 3’ एक्सोन्यूक्लिएस गतिविधि द्वारा आरएनए प्राइमर के न्यूक्लियोटाइड्स को हटा देता है और उन्हें अपनी 5’ → 3’ पॉलिमराइज़ेशन गतिविधि द्वारा पूरक डीएनए न्यूक्लियोटाइड्स से प्रतिस्थापित करता है। डीएनए लाइगेज़ ओकाज़ाकी खंडों को आपस में फॉस्फोडाइएस्टर बॉन्ड्स बनाकर जोड़ता है।
समापन प्रक्रिया के सटीक विवरण स्पष्ट नहीं हैं, लेकिन यह ज्ञात है कि वृत्ताकार डीएनए अणु पर मूल प्रतिकृतिकरण बिंदु के लगभग विपरीत एक विशिष्ट समापन स्थल होता है। प्रतिकृतिकरण तब समाप्त होता है जब दो प्रतिकृतिकरण कांटे आपस में मिलते हैं (चित्र 7.14)। एक समापन प्रोटीन समापन स्थल से बंधता है और हेलिकेज़ की गति को रोकता है, जिससे प्रतिकृतिकरण कांटा रुक जाता है और आगे का डीएनए प्रतिकृतिकरण रुक जाता है।
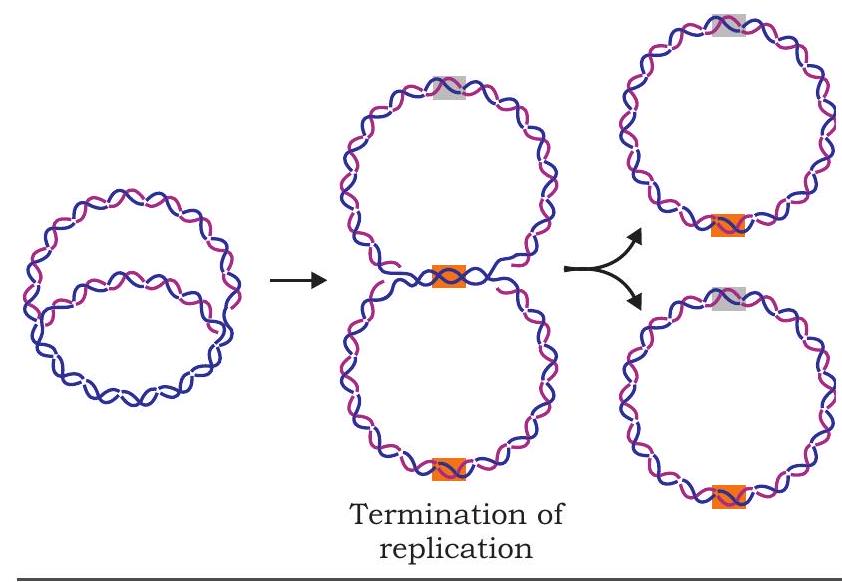
चित्र 7.14: प्रतिकृतिकरण का समापन
यूकैरियोटिक डीएनए प्रतिकृतिकरण कई मायनों में जीवाणु प्रतिकृतिकरण से मिलता-जुलता है। यूकैरियोट्स में कई गुणसूत्र होते हैं जिनमें रेखीय, द्वि-स्त्रंदीय, लंबे डीएनए अणु होते हैं। एकल उद्गम के बजाय, प्रत्येक यूकैरियोटिक गुणसूत्र में कई उद्गम होते हैं और प्रतिकृतिकरण कांटे द्विदिश होते हैं। सभी उद्गमों से एक साथ प्रतिकृतिकरण प्रारंभ हो सकता है। यूकैरियोट्स में डीएनए प्रतिकृतिकरण और मरम्मत में संलग्न कई प्रकार की डीएनए पॉलिमरेज़ पाई जाती हैं (चित्र 7.15)।
चित्र 7.15: यूकैरियोटिक गुणसूत्रों में कई उद्गम बिंदु होते हैं
7.4 जीन अभिव्यक्ति
जैसा कि हम पहले ही अध्ययन कर चुके हैं कि जीन डीएनए का एक खंड है जो किसी लक्षण की अभिव्यक्ति के लिए जैविक सूचना वहन करता है। जीन को समझने की एक बड़ी चुनौती यह है कि जीन अभिव्यक्त कैसे होता है। दूसरे शब्दों में, बहु-न्यूक्लियोटाइड श्रृंखला में न्यूक्लियोटाइडों की रेखीय अनुक्रम के रूप में यह सूचना, पॉलीपेप्टाइड श्रृंखला में अमीनो अम्लों की रेखीय अनुक्रम में कैसे परिवर्तित होती है?
प्रत्येक जीव, जीवाणु से लेकर मानव तक, जीन अभिव्यक्ति की एक ही मूलभूत प्रक्रिया रखता है (चित्र 7.16)। डीएनए की चार आधारों की अनुक्रम में संग्रहित सूचना अमीनो अम्लों को सही क्रम में जोड़ने का निर्देश देती है ताकि उस प्रोटीन का निर्माण हो जिसके लिए दिया गया डीएनए अनुक्रम उत्तरदायी है। एक जीव द्वारा प्राप्त किया गया डीएनए विशिष्ट प्रोटीनों के संश्लेषण का निर्देश देकर विशिष्ट लक्षणों को उत्पन्न करता है। दूसरे शब्दों में, प्रोटीन जीन प्रकार और लक्षण प्रकार के बीच की कड़ी हैं। डीएनए से प्रोटीन तक सूचना का यह एकदिशीय प्रवाह दो चरणों से होता है, अर्थात् प्रतिलेखन और अनुवाद, और इसे केन्द्रीय सिद्धांत कहा जाता है।
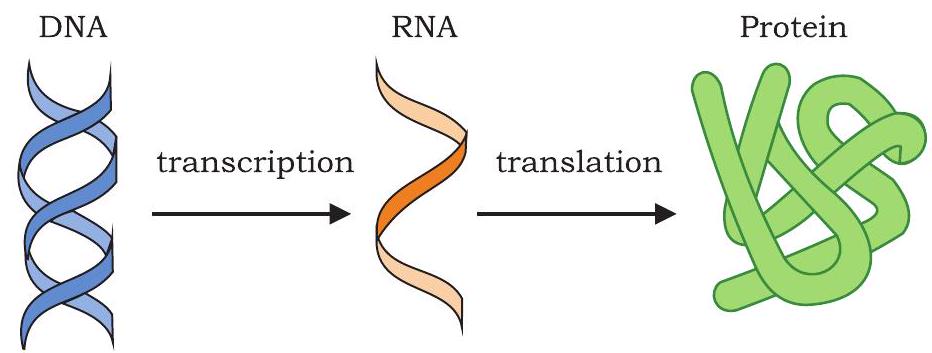
चित्र 7.16: केन्द्रीय सिद्धांत - सूचना का एकदिशीय प्रवाह
$\text{DNA}\xrightarrow []{\text{प्रतिलेखन}} \text{RNA} \xrightarrow []{\text{अनुवाद}} \text{पॉलीपेप्टाइड श्रृंखला}$
प्रतिलेखन के दौरान आनुवंशिक सूचना DNA से mRNA में स्थानांतरित होती है। केंद्रीय सिद्धांत के दूसरे चरण, अर्थात् अनुवाद में, सूचना mRNA से पॉलीपेप्टाइड श्रृंखला में स्थानांतरित होती है। कोशिका जीन जो DNA में उपस्थित है और वह पेप्टाइड जिसे वह कोडित करता है, के बीच mRNA को मध्यवर्ती बनाना क्यों चाहेगी? सबसे पहले, जीन में उपस्थित सूचना को DNA की एक प्रति से कई प्रतियों वाली mRNA बनाकर प्रवर्धित किया जा सकता है। दूसरा, यूकैरियोट्स में DNA केंद्रक के अंदर उपस्थित होता है और राइबोसोम, जो प्रोटीन के कारखाने हैं, कोशिकाद्रव्य में उपस्थित होते हैं। एक बार जब mRNA DNA स्ट्रैंड (जीन) की पूरक प्रति के रूप में संश्लेषित हो जाती है, तो वह कोशिकाद्रव्य में चली जाती है। कोशिकाद्रव्य में, यह राइबोसोम में पॉलीपेप्टाइड श्रृंखला के संश्लेषण के लिए एक टेम्पलेट के रूप में कार्य करती है (चित्र 7.17)।
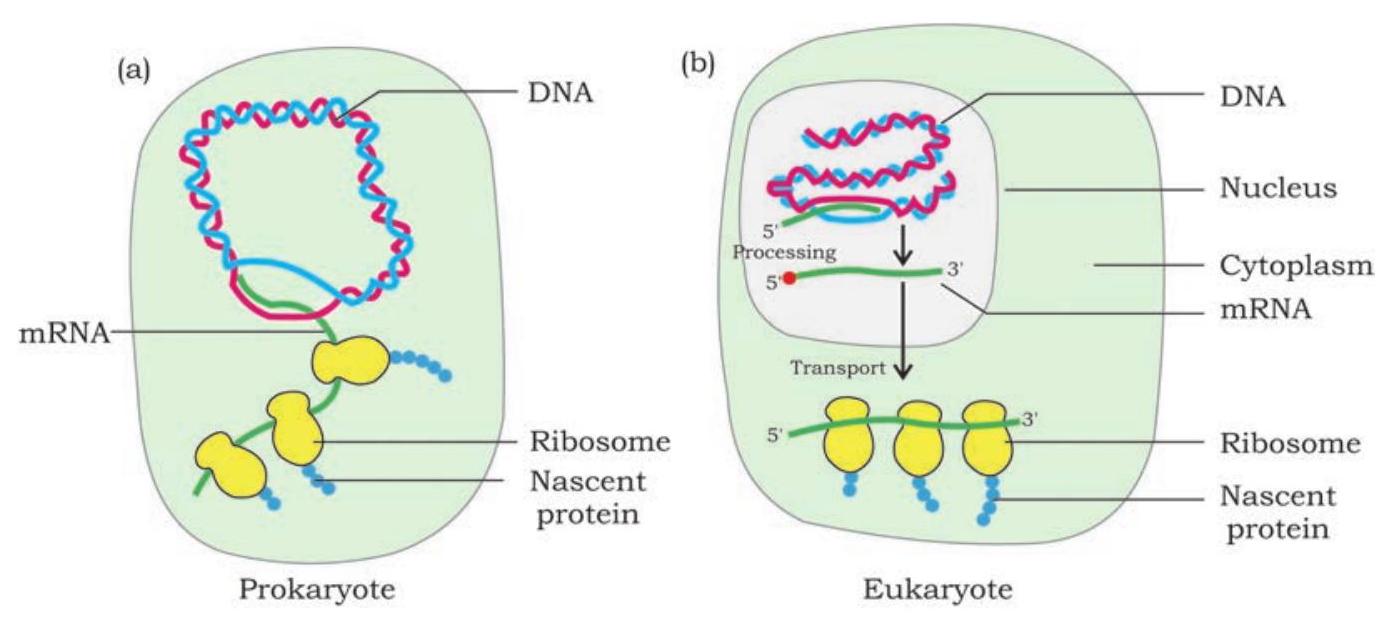
चित्र 7.17: प्रोकैरियोट्स और यूकैरियोट्स में जीन अभिव्यक्ति
कुछ वायरसों में, जैसे रेट्रोवायरस, आनुवंशिक पदार्थ DNA के बजाय RNA होता है। आनुवंशिक RNA में उपस्थित सूचना एकल स्ट्रैंड वाली पूरक DNA स्ट्रैंड में स्थानांतरित होती है जिसे फिर द्विस्ट्रैंड DNA में परिवर्तित किया जाता है। इस प्रक्रिया को रिवर्स ट्रांसक्रिप्शन कहा जाता है।
DNA के निर्माण के बाद, आनुवंशिक सूचना mRNA में और फिर पॉलीपेप्टाइड श्रृंखला में प्रवाहित होती है (चित्र 7.18)।
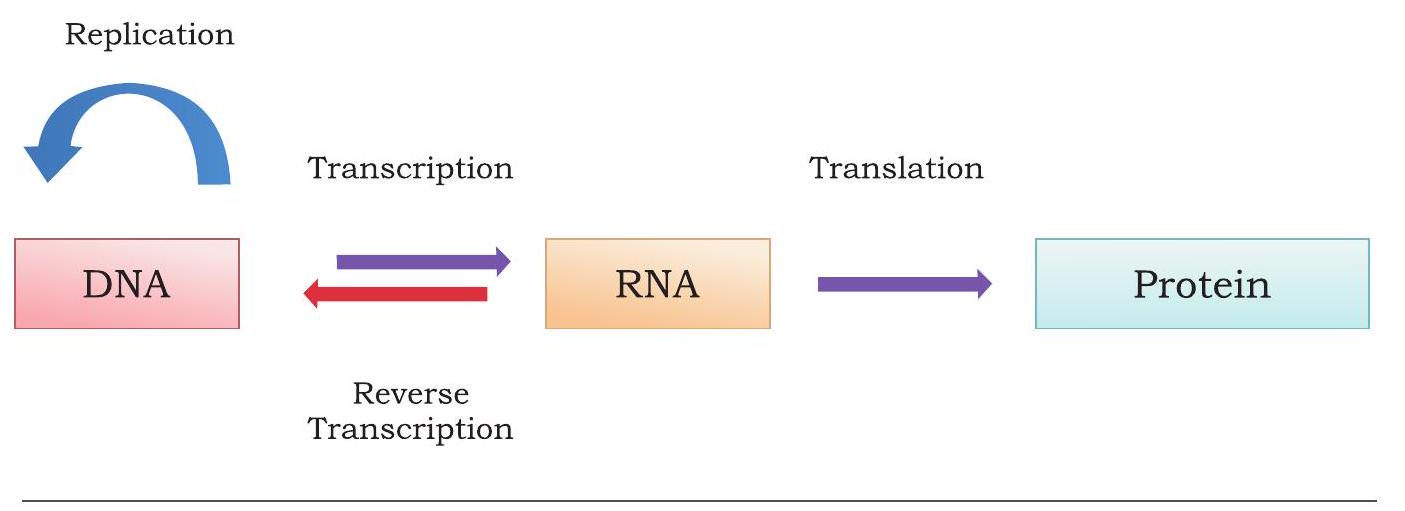
चित्र 7.18: आण्विक जीव विज्ञान के केंद्रीय सिद्धांत का उलट
7.4.1 ट्रांसक्रिप्शन
सभी प्रकार की कोशिकीय आरएनए जैसे मैसेंजर आरएनए (mRNA), ट्रांसफर आरएनए (tRNA), राइबोसोमल आरएनए (rRNA) डीएनए टेम्पलेट पर संश्लेषित होते हैं। डीएनए पर समपूरक आरएनए पॉलिन्यूक्लियोटाइड श्रृंखला का संश्लेषण ट्रांसक्रिप्शन कहलाता है। ट्रांसक्रिप्शन कई मायनों में प्रतिकृतिकरण प्रक्रिया के समान होता है। एक मौलिक अंतर यह है कि प्रतिकृतिकरण के दौरान संपूर्ण डीएनए टेम्पलेट को डीएनए अणु में कॉपी किया जाता है, लेकिन ट्रांसक्रिप्शन के दौरान डीएनए के केवल छोटे भागों को आरएनए में कॉपी किया जाता है। डीएनए स्ट्रैंड जिसके बेस अनुक्रम ट्रांस्क्राइब किए गए आरएनए के समान होता है (डीएनए में $\mathrm{T}$ के स्थान पर आरएनए में $\mathrm{U}$ को छोड़कर) जो आनुवंशिक सूचना वहन करता है, उसे सेंस या कोडिंग स्ट्रैंड कहा जाता है। दूसरा डीएनए स्ट्रैंड जिस पर आरएनए ट्रांस्क्राइब होता है और जिसका न्यूक्लियोटाइड अनुक्रम ट्रांस्क्राइब किए गए आरएनए के पूरक होता है, उसे टेम्पलेट या नॉन-कोडिंग स्ट्रैंड कहा जाता है (चित्र 7.20)। इस प्रकार, एक जीन के भीतर, न्यूक्लियोटाइड स्ट्रैंडों में से केवल एक ही सामान्यतः आरएनए में ट्रांस्क्राइब होता है।
7.4.2 आरएनए पॉलिमरेज
प्रोकैरियोट्स और यूकैरियोट्स दोनों में ट्रांसक्रिप्शन के लिए उत्तरदायी एंजाइम DNA-निर्भर RNA पॉलिमरेज है। प्रोकैरियोट्स में RNA पॉलिमरेज का एक ही प्रकार होता है जो सभी प्रकार के RNA (mRNA, rRNA और tRNA) के ट्रांसक्रिप्शन को उत्प्रेरित करता है। यूकैरियोट्स में RNA पॉलिमरेज के तीन भिन्न प्रकार मौजूद होते हैं। RNA पॉलिमरेज I, 28S, $18 \mathrm{~S}$ और $5.8 \mathrm{~S}$ का ट्रांसक्रिप्शन करता है; RNA पॉलिमरेज II, hnRNA (विषम केंद्रक RNA, mRNA का अग्रद्रव्य) का ट्रांसक्रिप्शन करता है और RNA पॉलिमरेज III, tRNA और 5S rRNA का ट्रांसक्रिप्शन करता है। RNA पॉलिमरेज एंजाइम पॉलीन्यूक्लियोटाइड श्रृंखला की संश्लेषण शुरू करने के लिए प्राइमर की आवश्यकता नहीं रखता और राइबोन्यूक्लियोसाइड ट्राइफॉस्फेट्स (rNTPs) को आधार बनाकर नई श्रृंखला को $5^{\prime} \rightarrow 3^{\prime}$ दिशा में संश्लेषित करता है।

चित्र 7.19: प्रोकैरियोटिक RNA पॉलिमरेज
प्रोकैरियोटिक RNA पॉलिमरेज एक बड़ा एंजाइम है जिसमें दो α, एक $\beta$ और एक $\beta$’ उपइकाई होती हैं (चित्र 7.19)। ये उपइकाइयाँ मिलकर कोर एंजाइम $\left(\alpha_{2}, \beta, \beta^{\prime}\right)$ बनाती हैं। जब कोर एंजाइम एक सिग्मा कारक से जुड़ता है तो यह होलोएंजाइम $\left(\alpha_{2}, \beta, \beta^{\prime}, \sigma\right)$ बनाता है।
7.4.3 ट्रांसक्रिप्शन इकाई
जिस डीएनए के हिस्से पर आरएनए का लेखन होता है उसे लेखन इकाई कहा जाता है। आरएनए पॉलिमरेज़ एक लेखन इकाई को कैसे पहचानता है? यह कैसे जानता है कि किस डीएनए स्ट्रैंड की प्रतिलिपि बनानी है और कहाँ से शुरू करना है और कहाँ रुकना है? प्रत्येक लेखन इकाई में एक प्रारंभ स्थल होता है जहाँ से लेखन आरंभ होता है और एक विरामक स्थल जहाँ लेखन समाप्त होना है (चित्र 7.20)। प्रारंभ स्थल के ऊपर की ओर एक डीएनए अनुक्रम होता है जिसे प्रमोटर कहा जाता है, जिसे आरएनए पॉलिमरेज़ पहचानता है और उससे जुड़कर लेखन प्रारंभ करता है। पॉलिमरेज़ के लिए बंधन स्थल देने के अतिरिक्त, प्रमोटर यह भी बताता है कि संश्लेषण कहाँ से शुरू करना है और किस दिशा में आगे बढ़ना है। प्रमोटर के भीतर सबसे सामान्य सहमति अनुक्रम TATAAT होता है जिसे बैक्टीरिया में प्रिब्नो बॉक्स कहा जाता है जहाँ डुप्लेक्स डीएनए का प्रारंभिक पिघलन होता है।
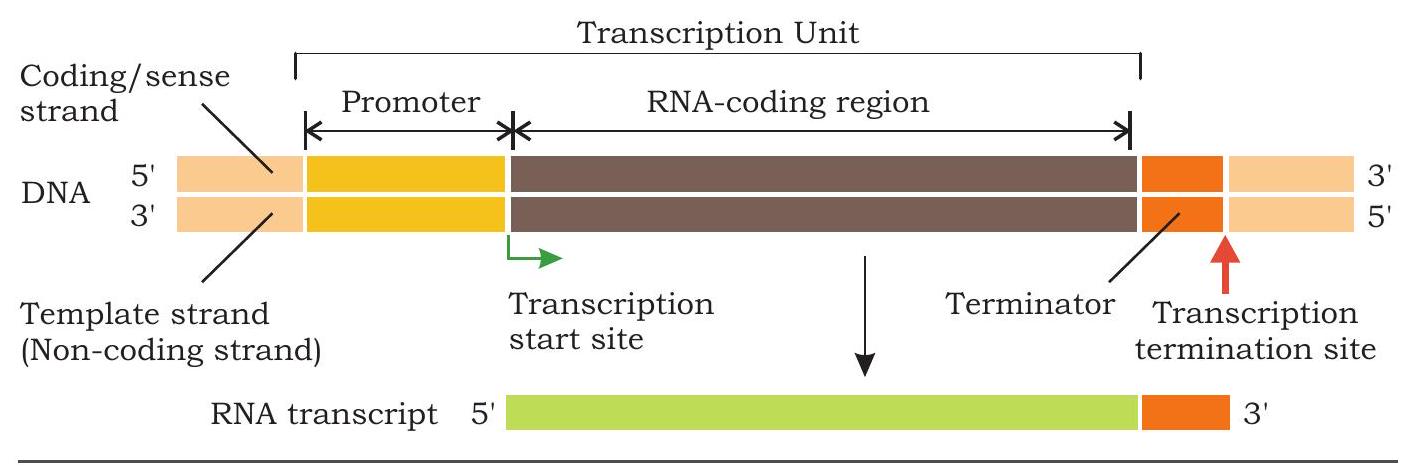
चित्र 7.20: एक लेखन इकाई
7.4.4 प्रारंभ
प्रमोटर से RNA पॉलिमरेज़ का बंधन ट्रांसक्रिप्शन का पहला चरण है। बैक्टीरिया में, RNA पॉलिमरेज़ का सिग्मा उपइकाई प्रमोटर को पहचानता है और उससे बंधता है। एक बार प्रमोटर से बंध जाने के बाद, RNA पॉलिमरेज़ होलोएंजाइम TATAAT अनुक्रम पर DNA हेलिक्स को खोलना शुरू करता है। यह लगभग 17 बेस जोड़ों लंबे DNA खंड को खोलता है। एक RNA अणु के संश्लेषण को शुरू करने के लिए, RNA पॉलिमरेज़ आने वाले राइबोन्यूक्लियोसाइड ट्राइफॉस्फेट के बेस को DNA टेम्पलेट स्ट्रैंड पर प्रारंभ स्थल के पूरक बेस से जोड़ता है। RNA अणु के 5’ सिरे के संश्लेषण को प्रारंभ करने के लिए कोई प्राइमर आवश्यक नहीं होता है। टेम्पलेट स्ट्रैंड के दूसरे न्यूक्लियोटाइड के पूरक अगला राइबोन्यूक्लियोसाइड ट्राइफॉस्फेट पहले RNA न्यूक्लियोटाइड के $3^{\prime} \mathrm{OH}$ सिरे पर RNA पॉलिमरेज़ द्वारा उत्प्रेरित फॉस्फोडाइएस्टर बंध द्वारा जोड़ा जाता है। इस दौरान, तीन फॉस्फेट समूहों में से दो ($\beta$ और $\gamma$) पाइरोफॉस्फेट के रूप में आने वाले राइबोन्यूक्लियोसाइड ट्राइफॉस्फेट से विद्यमान हो जाते हैं। प्रारंभ हो जाने के बाद सिग्मा उपइकाई सामान्यतः मुक्त हो जाता है।
7.4.5 विस्तार
वह क्षेत्र जिसमें RNA पॉलिमरेज़, DNA और बढ़ता हुआ RNA ट्रांसक्रिप्ट होता है, उसे ट्रांसक्रिप्शन बबल कहा जाता है क्योंकि इसमें DNA का स्थानीय रूप से खुला ‘बबल’ होता है (चित्र 7.21)।
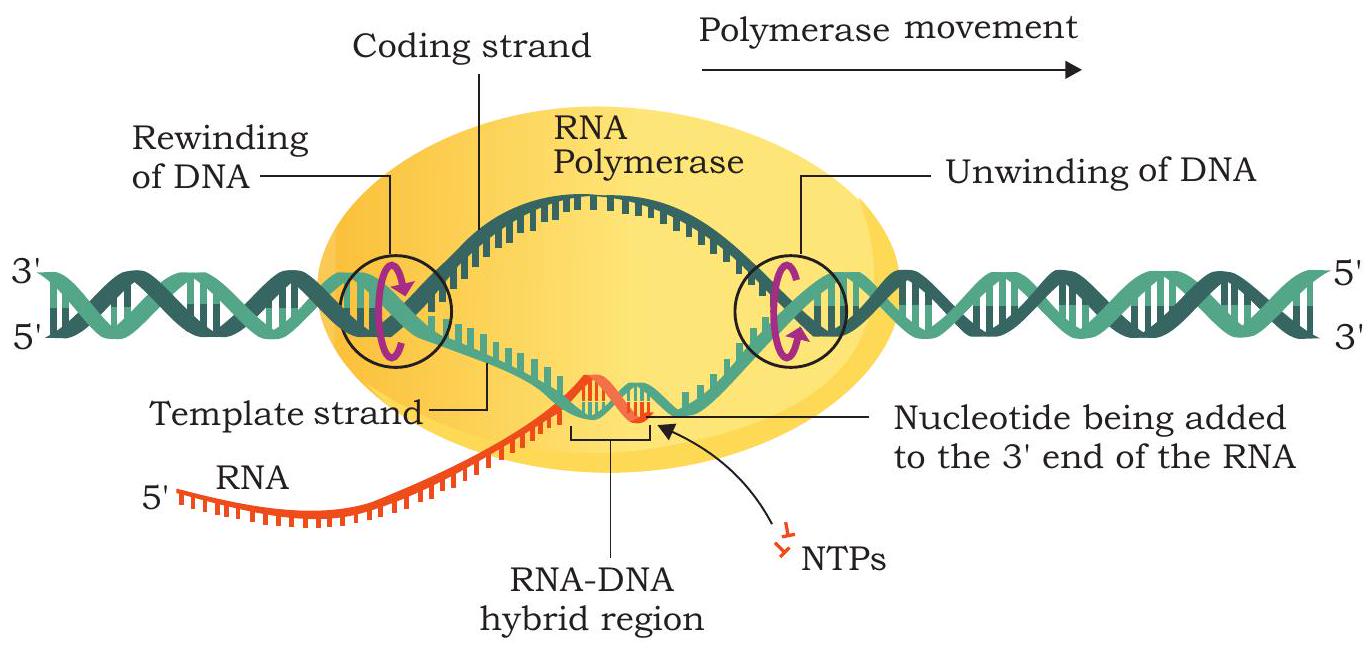
अनुवाद (हिन्दी):
चित्र 7.21: RNA पॉलिमरेज़ द्वारा RNA श्रृंखला का विस्तार
RNA पॉलिमरेज़ टेम्पलेट स्ट्रैंड के साथ क्रमिक रूप से आगे बढ़ता है और ट्रांसक्रिप्शन बबल के अग्रभाग पर DNA को अनवाइंड करता है। यह टेम्पलेट के अनुक्रम के अनुसार न्यूक्लियोटाइड्स को RNA अणु के 3′ OH सिरे से जोड़ता है (चित्र 7.22)। जैसे-जैसे RNA श्रृंखला का विस्तार होता रहता है, नई RNA अणु DNA टेम्पलेट से अलग होने लगती है और ट्रांसक्रिप्शन बबल के पीछे DNA द्विस्पिरल पुनः बन जाती है। 37 °C पर जीवाणु कोशिकाओं में ट्रांसक्रिप्शन की दर लगभग 40 न्यूक्लियोटाइड्स प्रति सेकंड होती है।

चित्र 7.22: बढ़ती हुई पॉलिन्यूक्लियोटाइड श्रृंखला के 3′ OH सिरे पर rNTP की जोड़ी
7.4.6 समापन
RNA श्रृंखला का विस्तार तब तक जारी रहता है जब तक RNA पॉलिमरेज़ DNA में टर्मिनेटर अनुक्रम तक नहीं पहुँच जाता। ट्रांसक्रिप्शन बबल के भीतर RNA-DNA हाइब्रिड हेलिक्स तब विघटित हो जाती है जब RNA पॉलिमरेज़ टर्मिनेटर अनुक्रम तक पहुँचता है। RNA पॉलिमरेज़ टेम्पलेट DNA से अलग हो जाता है, DNA की दोनों स्ट्रैंड्स पुनः लिपट जाती हैं और नव-संश्लेषित RNA श्रृंखला मुक्त हो जाती है। जीवाणुओं में ट्रांसक्रिप्शन समापन के दौरान कुछ स्थितियों में RNA पॉलिमरेज़ को रो प्रोटीन (रो-निर्भर समापन) की आवश्यकता होती है, जबकि अन्य स्थितियों में यह रो-निर्भर समापन द्वारा समाप्त होता है।
प्रोकैरियोट में, जीनों का एक समूह अक्सर एकल RNA अणु में ट्रांसक्राइब होता है, जिसे पॉलीसिस्ट्रोनिक RNA कहा जाता है। यह तब बनता है जब कई जीनों के एक समूह के अंत में एकल टर्मिनेशन अनुक्रम मौजूद होता है जो एक साथ ट्रांसक्राइब होते हैं। प्रोकैरियोट में, चूँकि mRNA को सक्रिय होने के लिए किसी प्रोसेसिंग की आवश्यकता नहीं होती, और चूँकि ट्रांसक्रिप्शन और अनुवाद दोनों ही कोशिकाद्रव्य में होते हैं, कई बार अनुवाद mRNA के पूरी तरह से ट्रांसक्राइब होने से पहले ही शुरू हो जाता है।
7.4.7 यूकैरियोट्स में ट्रांसक्रिप्शन
RNA पॉलिमरेज द्वारा ट्रांसक्रिप्शन की मूल प्रक्रिया यूकैरियोट्स में प्रोकैरियोट्स की तरह ही होती है। हालाँकि, ट्रांसक्रिप्शन की प्रक्रिया में प्रोकैरियोट्स और यूकैरियोट्स के बीच कई अंतर होते हैं। RNA पॉलिमरेज I, II और III द्वारा क्रमशः rRNA, mRNA और tRNA जीनों के प्रोमोटरों पर ट्रांसक्रिप्शन की उचित प्रारंभिकता के लिए ट्रांसक्रिप्शन कारक (transcription factors) के नाम से जाने जाने वाले कई सहायक कारकों की आवश्यकता होती है। यूकैरियोट्स में अधिकांश जीन विभाजित जीन होते हैं जिनमें एक्सॉन (कोडिंग क्षेत्र) के बीच इंट्रॉन (गैर-कोडिंग क्षेत्र) होते हैं। प्राथमिक ट्रांसक्रिप्ट्स में एक्सॉन और इंट्रॉन दोनों होते हैं। प्राथमिक ट्रांसक्रिप्ट्स, जिन्हें विषम केंद्रक RNA (hnRNA) कहा जाता है, न्यूक्लियस से कोशिकाद्रव्य में स्थानांतरित होने से पहले प्रोसेसिंग से गुजरते हैं, जिसे ट्रांसक्रिप्शन-पश्च संशोधन (Fig. 7.23) कहा जाता है।
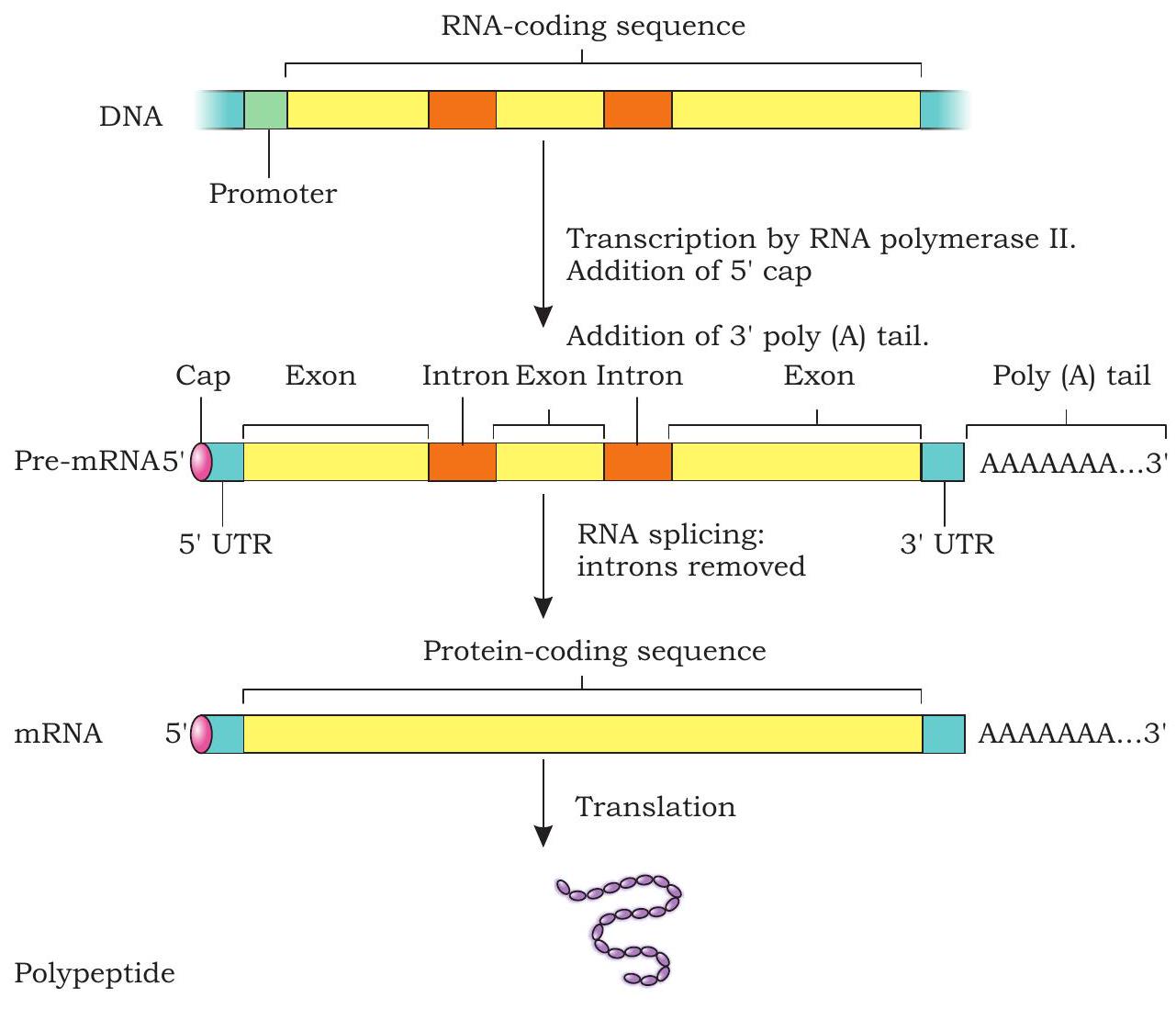
चित्र 7.23: प्री-mRNA के पश्च-प्रतिलेखन संशोधन
1. कैपिंग
यूकैरियोटिक प्री-mRNA में, पहला न्यूक्लियोटाइड 5’ सिरे पर एक प्यूरिन (A या G) होता है। प्री-mRNA के 5’ सिरे को GTP के जोड़ द्वारा संशोधित किया जाता है जिसे 5’ कैप कहा जाता है। GTP में मौजूद ग्वानिन में एक मेथिल समूह जोड़कर भी संशोधित किया जाता है जिसे अक्सर 5’ मेथिल G कैप कहा जाता है। यह कैप mRNA के 5’ सिरे को एक्सोन्यूक्लिएस द्वारा अपघटन से बचाता है।
2. स्प्लाइसिंग
प्राथमिक ट्रांसक्रिप्ट या प्री-mRNA में एक्सॉन और इंट्रॉन दोनों होते हैं। प्री-mRNA की प्रक्रिया के दौरान, इंट्रॉन को काटा जाता है और एक्सॉन को आपस में जोड़ा (स्प्लाइस) जाता है। इस प्रक्रिया को स्प्लाइसिंग कहा जाता है।
3. पॉली-A पूंछ
प्री-mRNA के प्रतिलेखन के बाद, $3^{\prime}$ सिरे पर एडेनिन न्यूक्लियोटाइड्स की एक श्रृंखला जोड़ी जाती है जिसे पॉली-A पूंछ या पॉली-एडेनिलेटेड पूंछ कहा जाता है। पॉली-A पूंछ mRNA की स्थिरता में भूमिका निभाती है क्योंकि यह उन्हें अपघटन से बचाती है।
7.5 जेनेटिक कोड
प्रतिकृति और प्रतिलेखन के मामले में, एक बहु-न्यूक्लिओटाइड श्रृंखला (टेम्पलेट स्ट्रैंड) की प्रतिलिपि बनाकर एक अन्य बहु-न्यूक्लिओटाइड स्ट्रैंड, अर्थात् क्रमशः एक डीएनए स्ट्रैंड या एक आरएनए स्ट्रैंड बनाया जाता है। ये प्रक्रियाएँ पूरकता के आधार पर समझना आसान हैं। परन्तु अनुवाद की प्रक्रिया में, आनुवंशिक सूचना न्यूक्लिओटाइडों के एक बहुलक (mRNA) से अमीनो अम्लों के एक बहुलक में स्थानांतरित होती है। न्यूक्लिओटाइडों और अमीनो अम्लों के बीच कोई पूरकता नहीं होती। प्रश्न उठता है कि mRNA में चार आधारों की क्रमिकता एक बहु-पेप्टाइड के अमीनो अम्ल क्रम को कैसे निर्दिष्ट करती है? प्रमाण सुझाते हैं कि न्यूक्लिओटाइड क्रम में एक सूक्ष्म परिवर्तन बहु-पेप्टाइडों के अमीनो अम्ल क्रम में परिवर्तन के साथ होता है। इसने एक आनुवंशिक कोड की कल्पना को जन्म दिया जो प्रोटीन संश्लेषण के दौरान अमीनो अम्ल क्रम को निर्देशित कर सकता है।
जीनेटिक कोड की कल्पना और उसकी व्याख्या वास्तव में चुनौतीपूर्ण थी क्योंकि इसमें भौतिकविदों, कार्बनिक रसायनज्ञों, जैवरसायनज्ञों और जेनेटिकिस्टों की सामूहिक भागीदारी और प्रयास आवश्यक थे। 1961 में फ्रांसिस क्रिक ने जीनेटिक कोड के अस्तित्व की कल्पना की और सुझाव दिया कि आनुवंशिक सूचना का वाहक आधार क्रम है। एक एकल अमीनो अम्ल को निर्दिष्ट करने के लिए कितने न्यूक्लियोटाइड आवश्यक हैं? चूँकि बीस भिन्न अमीनो अम्ल हैं और केवल चार भिन्न आधार हैं, यह तार्किक रूप से असंभव होगा कि प्रत्येक अमीनो अम्ल केवल एक न्यूक्लियोटाइड द्वारा निर्दिष्ट हो। इसी प्रकार, दो न्यूक्लियोटाइडों का संयोजन भी सोलह अमीनो अम्लों को निर्दिष्ट कर सकता है क्योंकि 16 कोडन बनेंगे। भौतिकविद जॉर्ज गैमो ने तर्क दिया कि चूँकि 4 आधार हैं और यदि उन्हें 20 अमीनो अम्लों के लिए कोड करना है, तो कोड आधारों के संयोजन से बना होना चाहिए। आगे उसने यह जोड़ा कि 20 अमीनो अम्लों का निर्माण करने के लिए 3 न्यूक्लियोटाइडों का एक जीनेटिक कोड बनना चाहिए। यह वास्तव में एक साहसिक कल्पना थी, क्योंकि $4^{3}$ का क्रमचय-संयोजन $4 \times 4 \times 4$ देता है जो 64 कोडन उत्पन्न करेगा, जो 20 भिन्न अमीनो अम्लों को निर्दिष्ट करने के लिए पर्याप्त से अधिक है।
अगला प्रमुख कदम यह निर्धारित करना था कि तीन-तीन न्यूक्लिओटाइड्स के कौन-से समूह कौन-से अमीनो अम्ल निर्दिष्ट करते हैं। 1961 में मार्शल निरेनबर्ग और जोहान मत्ताई द्वारा किए गए प्रयोग ने पहला कोडन पढ़ा। उन्होंने यूरेसिल को आधार के रूप में रखने वाले RNA न्यूक्लिओटाइड्स को जोड़कर एक कृत्रिम mRNA संश्लेषित किया। पॉली (U) RNA को एक टेस्ट ट्यूब में डाला गया जिसमें सभी बीस प्रकार के अमीनो अम्ल, राइबोसोम और प्रोटीन संश्लेषन के लिए आवश्यक अन्य घटक मौजूद थे। प्रत्येक टेस्ट ट्यूब में एक विशेष अमीनो अम्ल को रेडियोधर्मी बनाया गया। फेनिलएलानीन अमीनो अम्ल वाले एक टेस्ट ट्यूब में रेडियोधर्मी पॉलीपेप्टाइड श्रृंखला का पता चला। इस प्रकार निरेनबर्ग और मत्ताई ने निर्धारित किया कि mRNA कोडन UUU अमीनो अम्ल फेनिलएलानीन निर्दिष्ट करता है। पॉली (C) और पॉली (A) RNA का उपयोग करने वाले समान प्रयोगों के परिणामों ने दिखाया कि CCC प्रोलिन के लिए और AAA लाइसिन के लिए कोड करता है। निरेनबर्ग, मत्ताई, लेडर, ओचोआ और एच. जी. खोराना के प्रयोगों ने जेनेटिक कोड के सभी 64 कोडनों को पढ़ने में मदद की।
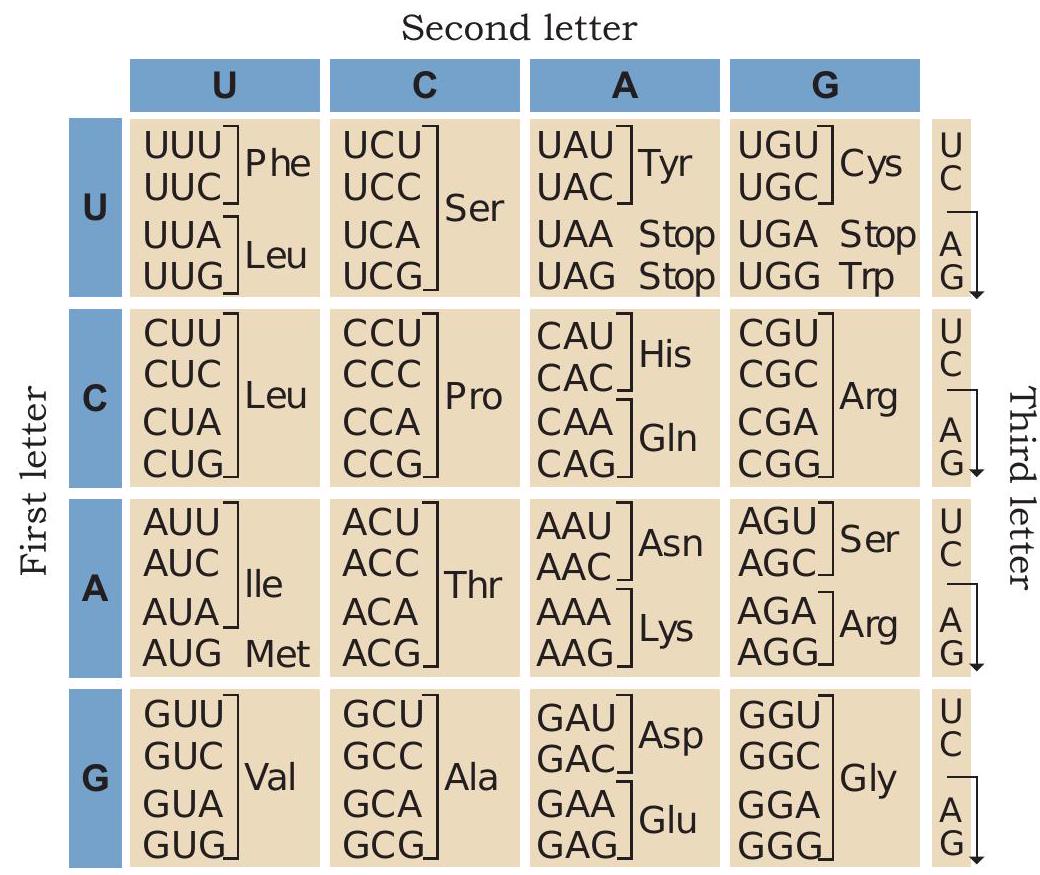
चित्र 7.24: जेनेटिक कोड
जेनेटिक कोड की प्रमुख विशेषताएँ इस प्रकार हैं:
1. कोडन त्रिक होते हैं और 64 कोडन होते हैं (चित्र 7.24)।
2. 64 कोडॉन में से 61 कोडॉन 20 अमीनो अम्लों के लिए कोड करते हैं। शेष तीन कोडॉन UAA, UAG और UGA किसी भी अमीनो अम्ल के लिए कोड नहीं करते और प्रोटीन संश्लेषण की समाप्ति का संकेत देने के लिए प्रयुक्त होते हैं।
3. AUG दोहरे कार्य करता है। यह मेथायोनीन के लिए कोड करता है और साथ ही प्रारंभक कोडॉन के रूप में भी कार्य करता है।
4. आनुवंशिक कोड असंदिग्ध है, अर्थात् एक कोडॉन केवल एक ही अमीनो अम्ल के लिए कोड करता है।
5. आनुवंशिक कोड अपभ्रष्ट है, जिसका अर्थ है कि प्रत्येक अमीनो अम्ल एक से अधिक कोडॉन द्वारा निर्दिष्ट किया जा सकता है। केवल मेथायोनीन और ट्रिप्टोफ़ान प्रत्येक एक-एक कोडॉन द्वारा एन्कोड किए जाते हैं।
6. आनुवंशिक कोड अतिव्यापी नहीं है। mRNA के साथ-साथ प्रत्येक नाइट्रोजनस क्षारक केवल एक ही कोडॉन का भाग होता है।
7. आनुवंशिक कोड सार्वत्रिक है; उदाहरण के लिए, बैक्टीरिया से लेकर मानव तक GUG वैलीन के लिए कोड करेगा। इस नियम के कुछ अपवाद माइटोकॉन्ड्रियल कोडॉन में और कुछ प्रोटोजोआ में पाए गए हैं।
7.6 अनुवाद
अब तक आपने सीखा है कि DNA में उपस्थित आनुवंशिक सूचना mRNA में ट्रांसक्राइब की जाती है। इस प्रकार, mRNA कोडॉन के रूप में विशिष्ट पॉलीपेप्टाइड श्रृंखलाओं के अमीनो अम्लों के क्रम के लिए आनुवंशिक सूचना ले जाता है। राइबोसोम में, कोडॉन की भाषा में उपस्थित सूचना को अमीनो अम्लों की भाषा में डिकोड किया जाता है जो एक पॉलीपेप्टाइड श्रृंखला बनाने के लिए जुड़ते हैं। mRNA पर राइबोसोम से बंधकर पॉलीपेप्टाइड श्रृंखला के संश्लेषण की प्रक्रिया को अनुवाद कहा जाता है। अनुवाद प्रक्रिया को चार चरणों में विभाजित किया जा सकता है:
(i) tRNA का आवेशन
(ii) अनुवाद का प्रारंभ
(iii) विस्तार
(iv) समाप्ति
tRNA का आवेशन
tRNA अणु राइबोसोम पर अमीनो अम्ल पहुँचाते हैं। tRNA के anticodon क्षेत्र में मौजूद तीन आधार mRNA के codon के विशिष्ट आधार को पहचानते हैं और उनके साथ युग्मित होते हैं (चित्र 7.25)। उदाहरण के लिए, mRNA का codon GAG ग्लूटामिक अम्ल की सूचना रखता है। tRNA का anticodon जो codon GAG के साथ हाइड्रोजन बंधन द्वारा आधार युग्मन करता है, वह CUC होता है और अपने दूसरे सिरे (3’ सिरा) पर ग्लूटामिक अम्ल ले जाता है। अनुवाद के दौरान, जब कोई mRNA अणु राइबोसोम से गुजरता है, तो GAG अनुवाद के लिए प्रस्तुत होने पर ग्लूटामिक अम्ल पॉलीपेप्टाइड श्रृंखला में जुड़ जाता है।
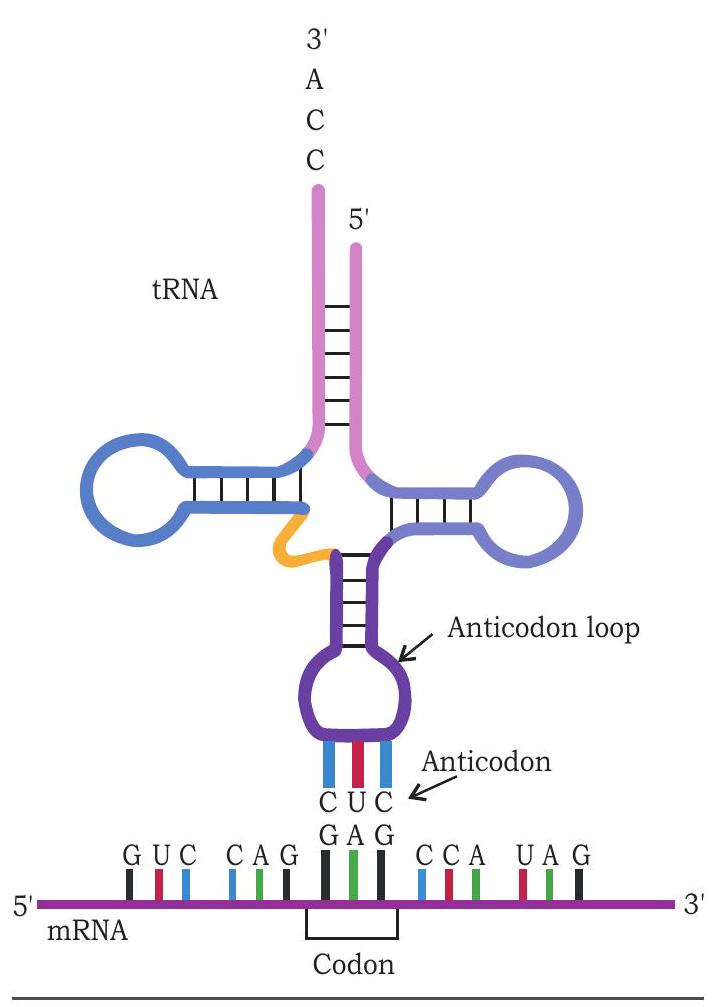
चित्र 7.25: tRNA का anticodon mRNA के codon को पहचानता है और उसके साथ आधार युग्मन करता है
प्रत्येक tRNA अणु एक विशिष्ट प्रकार का अमीनो अम्ल लेकर चलता है। tRNA अणुओं का नाम उस अमीनो अम्ल के अनुसार रखा जाता है जो वे ले जाते हैं। उदाहरण के लिए, मेथिओनिन ले जाने वाले tRNA को मेथिओनिल tRNA या tRNAmet कहा जाता है। इसी तरह, सीरिन ले जाने वाले tRNA को tRNAser कहा जाता है। जेनेटिक कोड में 61 कोडॉन 20 अमीनो अम्लों के लिए कोड करते हैं। 61 कोडॉनों के लिए कोशिका में 61 भिन्न tRNA अणुओं का होना चाहिए, जिनके एंटीकोडॉन भी भिन्न-भिन्न हों। परंतु, कोशिका में tRNA अणुओं की संख्या 61 से बहुत कम होती है। इसलिए, एक ही tRNA अणु का एंटीकोडॉन mRNA पर एक से अधिक कोडॉन को पहचान सकता है और उससे बेस-पेयरिंग कर सकता है। पर यह कैसे संभव है कि एक tRNA का एंटीकोडॉन एक से अधिक कोडॉन को पहचाने?
कोडॉन और एंटीकोडॉन के बीच बेस-पेयरिंग वॉट्सन-क्रिक नियम के अनुसार होती है, अर्थात् A का युग्म U से और G का युग्म C से होता है। पहली दो स्थितियों में यह युग्मन बिल्कुल सटीक होता है, जबकि कोडॉन की तीसरी स्थिति में यह लचीला होता है। तीसरे बेस पर इस असामान्य युग्मन को वॉबल युग्मन (Wobble hypothesis) कहा जाता है (चित्र 7.26)।
चित्र 7.26: कोडॉन की तीसरी स्थिति पर वॉबल युग्मन
अमीनो अम्लों को सर्वप्रथम ATP की उपस्थिति में aminoacyl-tRNA synthetase द्वारा सक्रियित किया जाता है और उन्हें उनके विशिष्ट tRNA अणुओं पर स्थानांतरित किया जाता है (चित्र 7.27)। एक कोशिका में 20 भिन्न aminoacyl-tRNA synthetase एंजाइम होते हैं, प्रत्येक 20 अमीनो अम्लों में से एक के लिए।
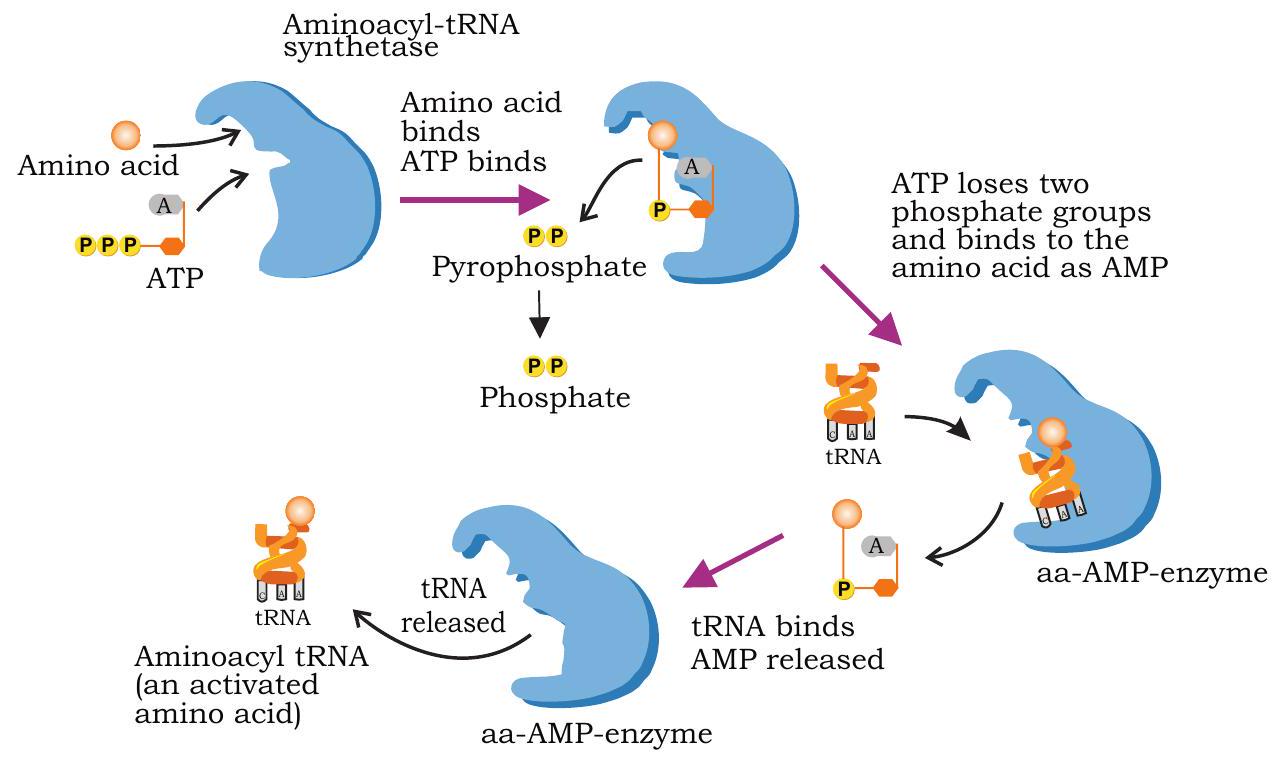
चित्र 7.27: अमीनो अम्ल का सक्रियीकरण और उसके विशिष्ट tRNA में स्थानांतरण जिससे aminoacyl-tRNA बनता है
प्रत्येक synthetase एक विशिष्ट अमीनो अम्ल को पहचानता है और उसे एक विशिष्ट tRNA अणु पर स्थानांतरित करता है। राइबोसोम में प्रोटीन संश्लेषण प्रारंभ होने से पहले अमीनो अम्ल सक्रिय होते हैं और उन्हें उनके उपयुक्त tRNA अणुओं पर स्थानांतरित किया जाता है। इसे tRNA का चार्जिंग कहा जाता है और इसमें दो चरण होते हैं।
पहले चरण में, अमीनो अम्ल aminoacyl tRNA synthetase की उपस्थिति में ATP के साथ अभिक्रिया करके aminoacyl-AMP और PPi उत्पन्न करता है। दूसरे चरण में, अमीनो अम्ल को tRNA पर स्थानांतरित किया जाता है। सक्रियित अमीनो अम्ल tRNA के 3′ OH सिरे पर उपस्थित टर्मिनल एडेनिन न्यूक्लियोटाइड के हाइड्रॉक्सिल समूह से जुड़ता है (CCA अनुक्रम)। चार्ज किया हुआ aminoacyl tRNA राइबोसोम में प्रवेश करता है।
$\text { अमीनो अम्ल }+ \text { ATP } \xrightarrow{\begin{array}{c} \text { Amino acyl } \\ \text { tRNA synthetase } \end{array}} \text { Aminoacyl-AMP }+\mathrm{PPi}$
$\text { एमिनोएसिल-एएमपी } + \text{ टी-आरएनए } \longrightarrow \text{ एमिनोएसिल-टी-आरएनए + एएमपी }$
अनुवाद का प्रारंभ
पिछली इकाई में हमने प्रोकैरियोटिक (70S) और यूकैरियोटिक (80S) राइबोसोम और उनके उपइकाइयों के बारे में अध्ययन किया है। राइबोसोम पर टी-आरएनए अणुओं के बंधन के लिए तीन स्थल होते हैं: A स्थल (एमिनोएसिल टी-आरएनए बंधन स्थल), P स्थल (पेप्टिडिल टी-आरएनए बंधन स्थल) और E स्थल (टी-आरएनए निर्गम स्थल)। अनुवाद के दौरान एमिनोएसिल-टी-आरएनए अणु एक के बाद एक A स्थल में प्रवेश करते हैं।
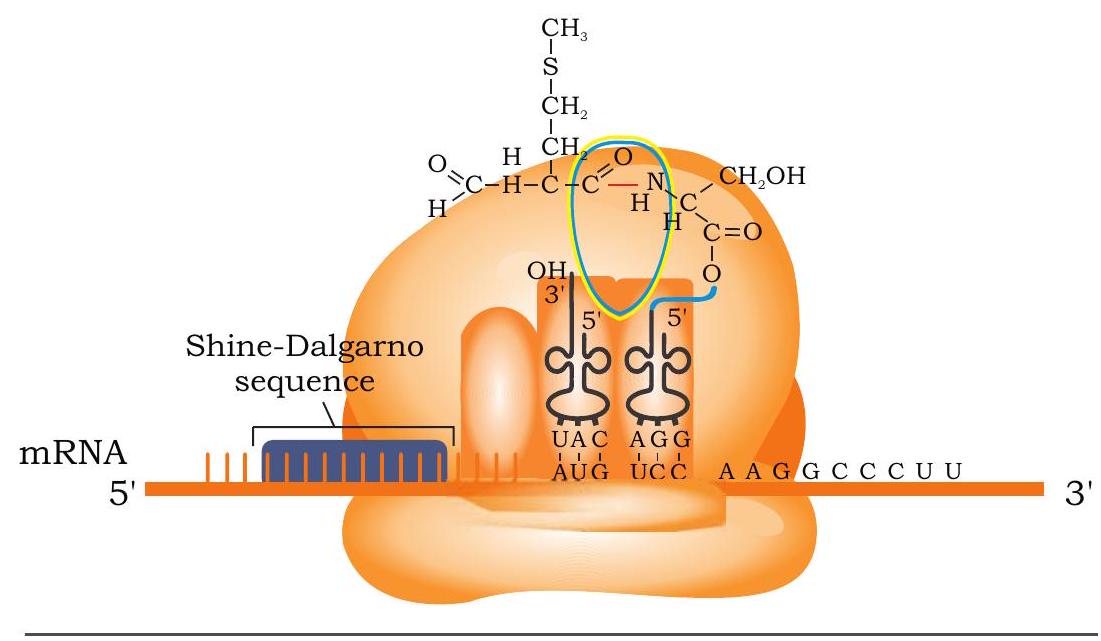
प्रारंभ के दौरान, प्रोकैरियोट्स में राइबोसोम की 30S उपइकाई mRNA के 5’ सिरे पर उपस्थित राइबोसोम बंधन अनुक्रम से बंधती है (चित्र 7.28)। परिणामस्वरूप, प्रारंभिक कोडन AUG (मेथियोनीन के लिए कोडित) P स्थल में स्थित हो जाता है।
प्रोकैरियोट्स में प्रारंभिक अमीनो एसिड मेथियोनीन फॉर्मिलेटेड होता है (फॉर्मिलेटेड मेथियोनीन - fMet) लेकिन यूकैरियोट्स में यह फॉर्मिलेटेड नहीं होता है। प्रोकैरियोट्स में प्रारंभिक चार्ज्ड tRNA या अमीनोएसिल-tRNA अणु fMet-tRNA $^{\text{fmet }}$ होता है (यूकैरियोट्स में Met-tRNA $^{\text{met }}$) जो P साइट में उपस्थित प्रारंभिक कोडन AUG से जुड़ता है। fMet-tRNA $^{\text{fmet }}$ का 3’ UAC 5’ एंटीकोडन mRNA के 5’ AUG 3’ कोडन के साथ बेस पेयरिंग करता है। 50S सबयूनिट तब 30S सबयूनिट से जुड़कर $70 \mathrm{~S}$ प्रारंभिक संकुल बनाता है। इस प्रारंभिक संकुल के निर्माण के लिए कुछ प्रोटीन कारकों जिन्हें प्रारंभिक कारक कहा जाता है और GTP की गतिविधि की आवश्यकता होती है। इसलिए, प्रारंभिक संकुल में, हमारे पास प्रारंभिक अमीनोएसिल-tRNA $(\text{fMet-tRNA} { }^{\text{fmet }})$ P साइट में होता है, जबकि A साइट खाली होता है, दूसरे अमीनोएसिल tRNA की आपूर्ति की प्रतीक्षा करता है।
यूकैरियोट्स में प्रारंभिक लगभग समान होता है लेकिन कुछ महत्वपूर्ण अंतर होते हैं। यूकैरियोटिक राइबोसोम का छोटा सबयूनिट (40S) प्रारंभिक कारकों की सहायता से कैप को पहचानता है, उससे जुड़ता है, और फिर mRNA के साथ आगे बढ़ता है जब तक कि यह प्रारंभिक AUG कोडन को नहीं ढूंढ लेता है।
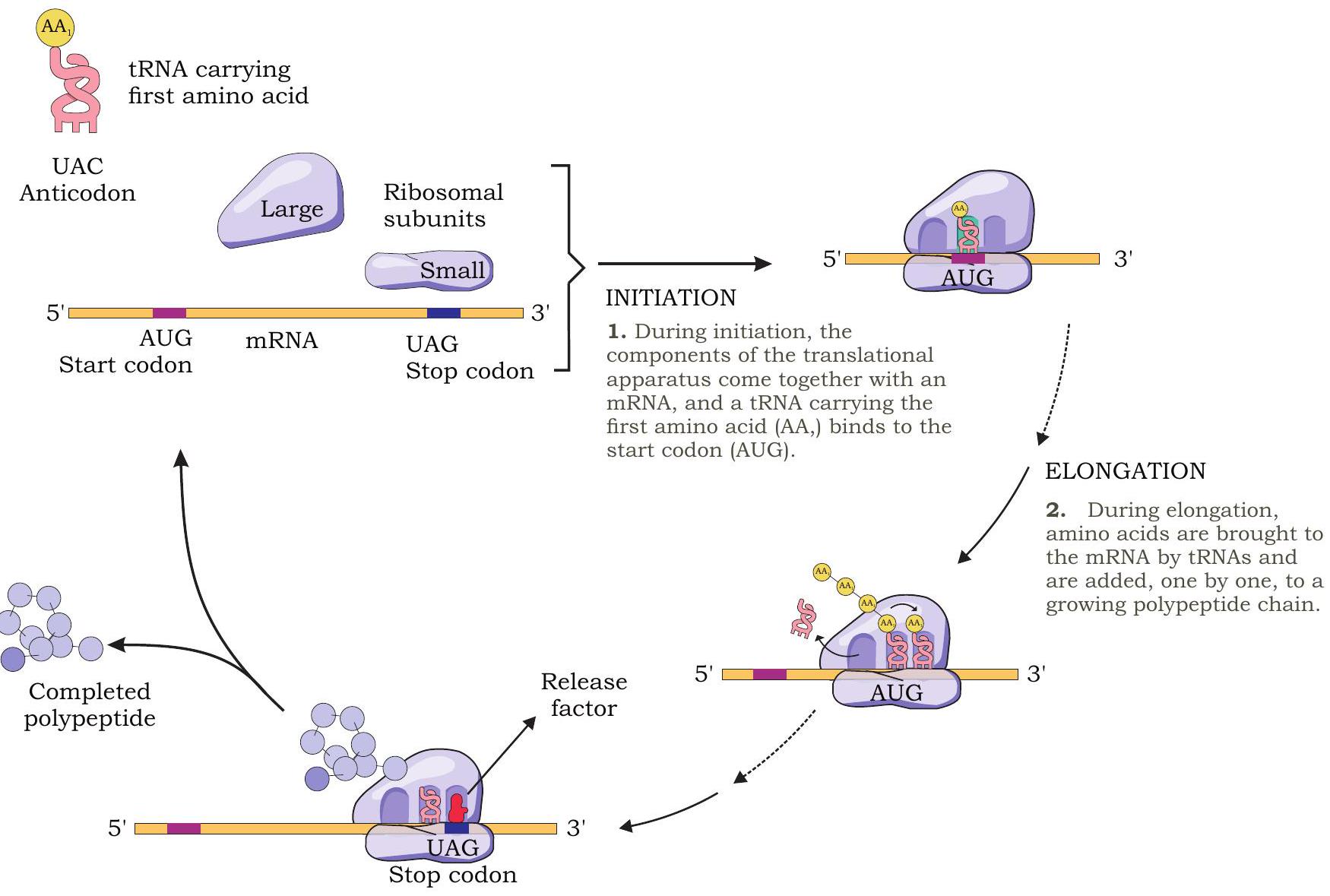
चित्र 7.28: प्रोकैरियोट्स में अनुवाद प्रक्रिया
विस्तार
प्रोटीन संश्लेषण में अगला चरण विस्तार (elongation) है, जिसमें अमीनो अम्लों को जोड़कर एक पॉलिपेप्टाइड श्रृंखला बनाई जाती है। प्रारंभिक संकुल (initiation complex) में, P स्थान पर फॉर्मिलेटेड मेथिओनिन (fMet-tRNAfmet) युक्त अमीनो एसिल-tRNA प्रोकैरियोट्स में और मेथिओनिन (Met-tRNAmet) युक्त tRNA यूकैरियोट्स में उपस्थित होता है (चित्र 7.29)। A स्थान खाली होता है। अब दूसरा अमीनोएसिल-tRNA उपयुक्त एंटीकोडन के साथ A स्थान में प्रवेश करता है जो mRNA कोडन के साथ बेस जोड़ता है। अब P और A स्थानों पर tRNA से जुड़े अमीनो अम्लों के बीच एक पेप्टाइड बंध बनता है। पेप्टाइड बंध P स्थान में प्रारंभिक tRNA से बंधे अमीनो अम्ल की कार्बोक्सिल समूह और A स्थान में tRNA से जुड़े अमीनो अम्ल के मुक्त अमीनो समूह के बीच बनता है। यह अभिक्रिया पेप्टिडिल ट्रांसफरेज एंजाइम द्वारा उत्प्रेरित होती है।
चित्र 7.29: प्रारंभिक अमीनो अम्ल (fMet) और दूसरे अमीनो अम्ल के बीच पेप्टाइड बंध का निर्माण
पेप्टाइड बंध के निर्माण से P स्थान में उपस्थित अमीनो अम्ल अपने tRNA से मुक्त हो जाता है। इस प्रकार A स्थान में एक डाइपेप्टिडिल-tRNA होता है और P स्थान में एक अनचार्ज्ड tRNA (अमीनो अम्ल रहित) होता है।
राइबोसोम अब mRNA के साथ $5^{\prime} \rightarrow 3^{\prime}$ दिशा में तीन न्यूक्लियोटाइड्स की दर से आगे बढ़ता है। इस गति को ट्रांसलोकेशन कहा जाता है। यह गति अनचार्ज्ड tRNA को $\mathrm{P}$ साइट से $\mathrm{E}$ साइट पर ले जाती है जहाँ से इसे राइबोसोम से बाहर निकाल दिया जाता है। बढ़ती हुई पॉलीपेप्टाइड चेन वाला पेप्टिडिल-tRNA A साइट से $\mathrm{P}$ साइट पर चला जाता है। राइबोसोम का A साइट अब फिर से खाली हो जाता है और mRNA का एक नया कोडन इस पर आ जाता है। यह अब अगले अमिनोएसिल-tRNA अणु को कोडन द्वारा निर्दिष्ट होकर प्राप्त करने के लिए तैयार हो जाता है। संपूर्ण प्रक्रिया दोहराई जाती है और पॉलीपेप्टाइड चेन का विस्तार होता है। विस्तार चरण के दौरान कई प्रोटीन कारक जिन्हें विस्तार कारक कहा जाता है और GTP शामिल होते हैं।
समापन
पॉलीपेप्टाइड चेन का विस्तार तब तक जारी रहता है जब तक कि mRNA पर कोई स्टॉप कोडन राइबोसोम के A साइट में प्रवेश नहीं कर जाता। तीन स्टॉप कोडन—UAA, UAG और UGA—किसी भी अमीनो अम्ल के लिए कोड नहीं करते। इन स्टॉप कोडनों के पूरक एंटीकोडन वाले कोई tRNA नहीं होते। जब समापन कोडन A साइट पर होता है तो कोई भी अमीनो अम्ल युक्त tRNA राइबोसोम के $\mathrm{A}$ साइट में प्रवेश नहीं करता। रिलीज कारक कहे जाने वाले प्रोटीन कारक स्टॉप कोडनों को पहचानते हैं और A साइट से बाइंड होते हैं (चित्र 7.30)।
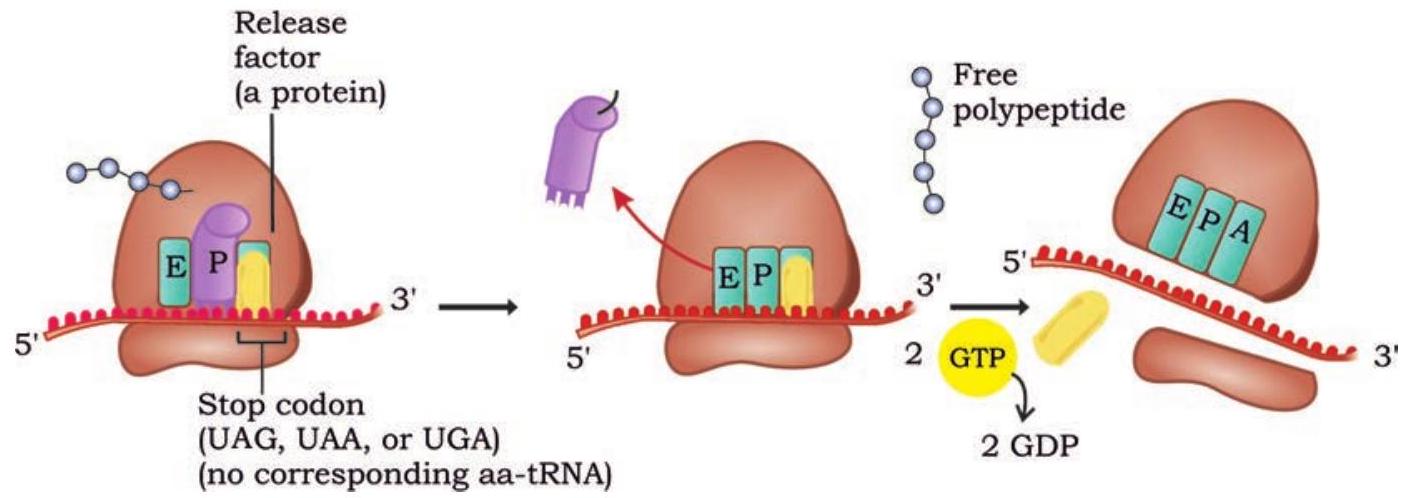
चित्र 7.30: रिलीज कारक स्टॉप कोडन को पहचानता है और ट्रांसलेशन प्रक्रिया को समाप्त करता है
रिलीज़ कारक तब पॉलीपेप्टाइड श्रृंखला को $\mathrm{P}$ साइट में tRNA से मुक्त करते हैं। अन्य प्रोटीन कारक $\mathrm{P}$ साइट से tRNA, राइबोसोम से mRNA और अंत में राइबोसोम के विघटन की रिलीज़ लाते हैं।
राइबोसोम में संश्लेषित प्रोटीन्स फिर परिपक्व प्रोटीन उत्पाद बनाने के लिए अनुवादोत्तर संशोधनों (PTMs) से गुजरते हैं। ऐसे संशोधन कई प्रकार के होते हैं और अधिकांशतः एंजाइमों द्वारा उत्प्रेरित होते हैं जो विशिष्ट प्रोटीनों में विशिष्ट लक्ष्य अनुक्रमों को पहचानते हैं।
पॉलीराइबोसोम
एक ही mRNA का एक साथ कई राइबोसोम द्वारा अनुवाद होता है जिससे पॉलीपेप्टाइड श्रृंखला की कई प्रतियां बनती हैं। जब पहला राइबोसोम mRNA से जुड़कर प्रारंभिक कोडोन से परे परिवहित हो जाता है, तब दूसरा राइबोसोम उसी mRNA से जुड़ जाता है, जिससे अंततः mRNA से जुड़े कई राइबोसोम बनते हैं जिन्हें पॉलीराइबोसोम कहा जाता है (चित्र 7.31)। ये प्रोकैरियोट्स और यूकैरियोट्स दोनों में पाए जाते हैं।
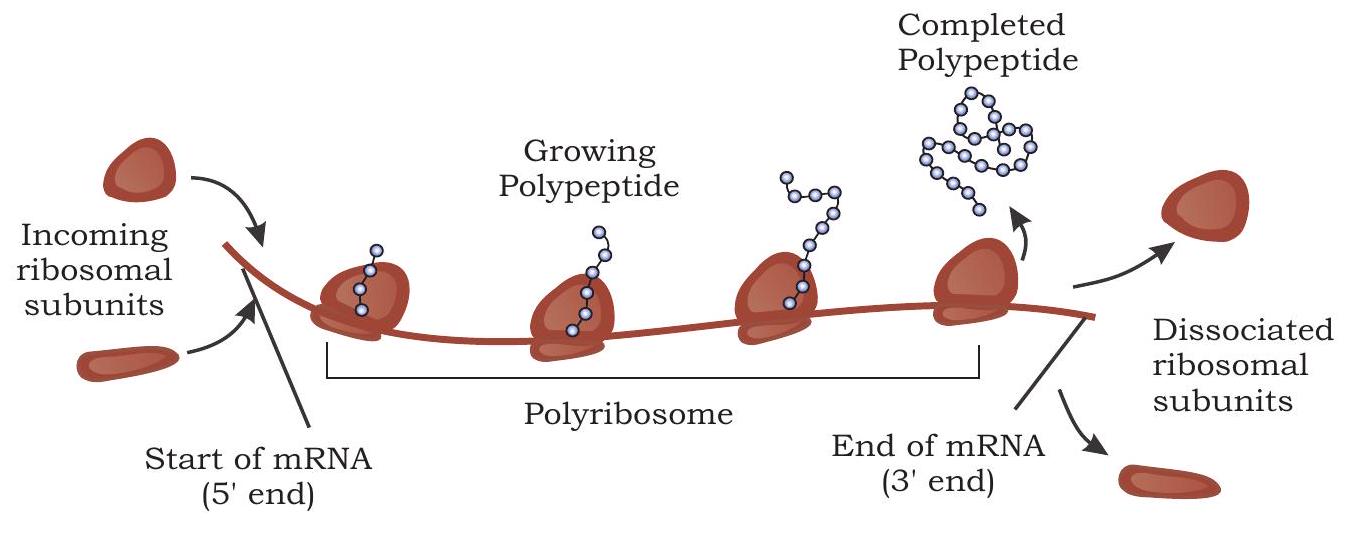
चित्र 7.31: अनुवाद के दौरान कई राइबोसोम एक mRNA से जुड़कर पॉलीराइबोसोम बनाते हैं
7.7 जीन उत्परिवर्तन
आपने यह बात समझ ली है कि किसी जीव में लक्षण या विशेषताएँ जीनों द्वारा नियंत्रित या विनियमित होती हैं, जो गुणसूत्रों की DNA का एक भाग होते हैं। लक्षण माता-पिता से संतान तक DNA या गुणसूत्रों पर मौजूद इन जीनों के माध्यम से कोडित सूचना के रूप में विश्वसनीय रूप से वंशानुगत होते हैं। सभी तंत्र और प्रक्रियाएँ जैसे DNA प्रतिकृतिकरण, ट्रांसक्रिप्शन और कोशिका विभाजन (माइटोसिस या मियोसिस) की प्रक्रिया के दौरान गुणसूत्रों का वितरण आदि विशिष्ट एंजाइम के नियंत्रण में अत्यंत सटीक और निश्चित होते हैं। फिर भी इन अणुीय प्रक्रियाओं के दौरान कुछ त्रुटि होने की संभावनाएँ होती हैं जिससे गुणसूत्रीय संरचना और जीनों को वहन करने वाली DNA की अणुीय संरचना में परिवर्तन हो सकते हैं। इन परिवर्तनों को व्यापक रूप से उत्परिवर्तन या आनुवंशिक पदार्थ में आकस्मिक परिवर्तन के रूप में वर्गीकृत किया जाता है। इस प्रकार, अब यह स्पष्ट है कि आनुवंशिक सूचना के वाहक, अर्थात् DNA या गुणसूत्र दोनों में ही सोमैटिक तथा जर्म कोशिका दोनों में परिवर्तन हो सकते हैं। हालाँकि, ऐसा परिवर्तन जब तक जर्म कोशिका में न हो और संतान में वंशानुगत न हो, तब तक इसका कोई महत्व नहीं होता।
आइए अब परिवर्तन या संशोधनों की विभिन्न श्रेणियों की जांच करें। यूकैरियोट्स के बीच, जर्म कोशिका में उपस्थित गुणसूत्र माता-पिता से संतान तक आनुवंशिक सूचना वहन करते हैं। इसलिए, गुणसूत्र की संरचना में कोई भी परिवर्तन (गुणसूत्रीय विसंगति) या गुणसूत्रों की कुल संख्या में परिवर्तन (प्लॉइडी) को गुणसूत्रीय उत्परिवर्तन की श्रेणी में रखा जाता है। विसंगति या तो किसी भाग के ह्रास के कारण या गुणसूत्र में किसी भाग के योग के कारण हो सकती है। गुणसूत्रीय खंड का पुनः व्यवस्थापन चाहे एक गुणसूत्र के भीतर हो या दो गुणसूत्रों के बीच, उसे भी गुणसूत्रीय विसंगति की श्रेणी में रखा जाता है। कई बाहरी कारक जैसे आयनकारी विकिरण या कुछ रसायन ऐसी विसंगति को उत्पन्न कर सकते हैं जिन्हें उत्परिवर्तक कहा जाता है। आप समझेंगे कि ऐसे सभी पुनः व्यवस्थापनों की पहचान या तो विशिष्ट गुणसूत्र रंग तकनीक जिसे बैंडिंग कहा जाता है या फ्लोरेसेंस इन सिटू हाइब्रिडाइजेशन (FISH) द्वारा की जा सकती है, जिसे आप इकाई V में बाद में पढ़ेंगे। इसी प्रकार, कुछ असाधारण परिस्थितियाँ हो सकती हैं जिनमें गुणसूत्रों की कुल संख्या (जो एक पीढ़ी से दूसरी पीढ़ी तक स्थिर रहती है) में परिवर्तन हो सकता है या तो वृद्धि के कारण या कमी के कारण एक या दोनों समजात गुणसूत्रों द्वारा। ऐसी स्थिति को अर्धप्लॉइडी की श्रेणी में वर्णित किया जाता है और आप बाद में पाएंगे कि यह मानवों में प्रेक्षित विभिन्न प्रकार की आनुवंशिक सिंड्रोम के लिए उत्तरदायी है। इसी प्रकार, संख्या में परिवर्तन संपूर्ण हेप्लॉइड समुच्चय के गुणन के कारण हो सकता है जिससे संख्या $3n, 4n$ या इससे भी अधिक हो जाती है जिसे पॉलीप्लॉइडी कहा जाता है।
अब आप सोच रहे होंगे कि यदि गुणसूत्र स्तर पर ऐसा परिवर्तन हो सकता है या उसे उत्पन्न किया जा सकता है, तो सम्भव है कि आनुवंशिक पदार्थ, अर्थात् DNA या RNA में भी अणु स्तर पर परिवर्तन हो सकते हैं। जीवाणुओं में केवल एक वृत्ताकार DNA होता है या यूकैरियोट के प्रत्येक गुणसूत्र में DNA होता है और सभी माइटोसिस या मियोसिस से पहले प्रतिकृतिकरण नामक प्रक्रिया द्वारा अपनी प्रति बनाने की प्रक्रिया में संलग्न होते हैं। प्रतिकृतिकरण के दौरान या अन्य किसी कारण से होने वाली कोई त्रुटि किसी एक या अन्य जीन के आनुवंशिक कोड के पठन-ढाँचे को बदल सकती है और इस प्रकार कोड को बदल सकती है और इसलिए उस लक्षण को प्रभावित कर सकती है जिसे वह जीन कोडित करता है। आनुवंशिक पदार्थ में अणु स्तर पर ऐसा परिवर्तन जीन उत्परिवर्तन या बिन्दु उत्परिवर्तन के रूप में वर्गीकृत किया जाता है। सिकल सेल ऐनीमिया उत्परिवर्तन का एक ऐसा ही उदाहरण है जिसमें एक न्यूक्लियोटाइड के प्रतिस्थापन से मानव RBC में असामान्य सिकल हीमोग्लोबिन का निर्माण होता है और परिणामस्वरूप रोग उत्पन्न होता है। बाह्य कारक (उत्परिवर्तज), भौतिक (आयनकारी विकिरण, UV किरणें), रासायनिक या जैविक (विषाणु) जीन उत्परिवर्तन उत्पन्न कर सकते हैं। हम अपना ध्यान मुख्यतः जीन उत्परिवर्तन पर केन्द्रित करेंगे।
अब यह स्पष्ट हो गया है कि जीनेटिक पदार्थ, अर्थात् DNA (कुछ वायरस में RNA) में परिवर्तन आणविक प्रक्रियाओं के दौरान हो सकता है। यह देखा गया है कि अणु या प्रक्रिया की कुछ आंतरिक विशेषताएं DNA या जीन में परिवर्तन का कारण बन सकती हैं, जो अणु स्तर पर संरचनात्मक संगठन के दृष्टिकोण से होता है। इन परिवर्तनों को तीन अलग-अलग समूहों में वर्गीकृत किया जा सकता है, अर्थात् एक या कुछ न्यूक्लियोटाइड्स की जोड़, विलोपन और प्रतिस्थापन। इनमें से, दो प्रकार के परिवर्तन, अर्थात् जोड़ और विलोपन, DNA अणु पर न्यूक्लियोटाइड अनुक्रम के पूरे रीडिंग फ्रेम को बदल देते हैं। इस तरह के परिवर्तन के प्रभाव को इस तथ्य से समझा जा सकता है कि DNA की बदली हुई कोडिंग RNA ट्रांसक्रिप्शन के दौरान और अंततः पॉलीपेप्टाइड श्रृंखला संश्लेषण के दौरान इसकी अभिव्यक्ति को बदल सकती है। स्पष्ट है कि इस तरह से संशोधित जीन द्वारा संश्लेषित प्रोटीन सामान्य नहीं हो सकता है या विशिष्ट प्रोटीन का संश्लेषण भी नहीं हो सकता है।
जीन में परिवर्तन एकल न्यूक्लियोटाइड के प्रतिस्थापन या प्रतिस्थापन के कारण भी हो सकता है। न्यूक्लियोटाइड की प्रतिस्थापन तब हो सकती है जब एक प्यूरिन बेस को दूसरे प्यूरिन बेस से प्रतिस्थापित किया जाता है। पाइरीमिडिन बेस के साथ भी ऐसा ही हो सकता है। इस तरह के प्रतिस्थापन प्रकार के उत्परिवर्तन को संक्रमण (transition) कहा जाता है। इसी प्रकार, एक प्यूरिन बेस को पाइरीमिडिन बेस से या इसके विपरीत प्रतिस्थापित किया जा सकता है, जिसे ट्रांसवर्जन (transversion) कहा जाता है (चित्र 7.32)।
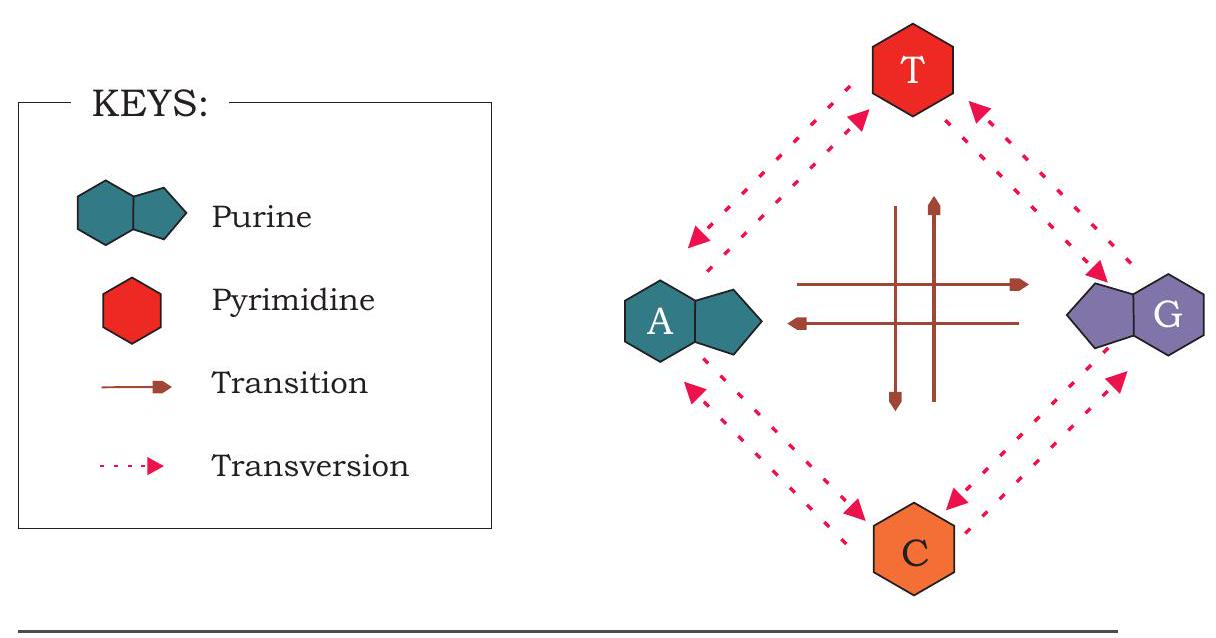
चित्र 7.32: विभिन्न प्रतिस्थापी उत्परिवर्तन
7.7.1 उत्परिवर्तन की आण्विक प्रक्रिया
जैसा कि पहले चर्चा की गई थी, उत्परिवर्तन स्वतः ही अणु के आंतरिक गुणों के कारण हो सकते हैं या वे बाहरी एजेंटों, जिन्हें उत्परिवर्तक कहा जाता है, द्वारा प्रेरित भी हो सकते हैं।
स्वतः उत्परिवर्तन- डीएनए न्यूक्लियोटाइड के रूप में आनुवंशिक सूचना वहन करता है। इन न्यूक्लियोटाइडों में कार्यात्मक समूह या तो $\mathrm{C}=\mathrm{O}$ या $\mathrm{C}-\mathrm{NH}_{2}$ रूप में उपस्थित होते हैं, जिन्हें सामान्यतः क्रमशः कीटो या अमीनो रूप कहा जाता है। हालांकि, नाइट्रोजिनस बेस के इन रूपों में हाइड्रोजन परमाणु अणु के एक परमाणु से दूसरे परमाणु में स्थानांतरित हो सकता है। ऐसी घटना को टॉटोमेरिक शिफ्ट कहा जाता है और यह नाइट्रोजिनस बेस की एक अस्थायी अवस्था उत्पन्न करती है जिसे या तो इनॉल (C - $\mathrm{OH}$) या इमिनो $(\mathrm{C}=\mathrm{NH})$ रूप कहा जाता है। नाइट्रोजिनस बेस के ये दुर्लभ टॉटोमेरिक रूप डीएनए अणु में अन्य न्यूक्लियोटाइड के साथ जोड़ने की बदली हुई संपत्ति रखते हैं। इसलिए, प्रतिकृतिकरण के समय, जब डीएनए में ग्वानिन का इमिनो रूप उपस्थित होता है, तो यह थाइमिन न्यूक्लियोटाइड के साथ पूरक युग्मन बनाता है जबकि सामान्यतः यह साइटोसिन के साथ युग्मित होता। अगले प्रतिकृतिकरण चक्र के दौरान, जहाँ थाइमिन गलत तरीके से जोड़ा गया है, वहाँ सामान्यतः साइटोसिन युग्मित हो सकता है जिससे जीन में $G \equiv C$ युग्म की जगह $\mathrm{A}=\mathrm{T}$ युग्म का प्रतिस्थापन होता है (चित्र 7.33)। हमने सिकल सेल एनीमिया के मामले में एकल बेस प्रतिस्थापन के परिणाम पर चर्चा की है।

चित्र 7.33: प्रतिस्थापन उत्परिवर्तन के स्वतः प्रेरण को दर्शाता है
प्रेरित उत्परिवर्तन- हरमन जे. मुलर द्वारा एक्स-किरण का उपयोग कर उत्परिवर्तन प्रेरित करने के प्रयोगों के बाद, विभिन्न बाह्य कारकों का उपयोग कर उत्परिवर्तन प्रेरित करने का एक नया क्षेत्र खुला। ये उत्परिवर्तज निम्न श्रेणियों में आते हैं:
-
भौतिक- एक्स-किरण, यूवी किरण आदि जैसी विकिरणें।
-
रासायनिक- मस्टर्ड गैस, एथिल मीथेन सल्फोनेट (EMS) जैसे एल्किलेटिंग एजेंट; 5-ब्रोमोयूरेसिल या 2-एमिनोप्यूरिन जैसे बेस एनालॉग; नाइट्रस अम्ल आदि डीएमिनेटिंग एजेंट।

चित्र 7.34: डीएनए प्रतिकृत्ति पर 5-ब्रोमोयूरेसिल का प्रभाव
बाह्य कारक द्वारा उत्परिवर्तन प्रेरण की क्रिया-विधि को समझने के लिए कुछ उदाहरणों से प्रयास करें। कोई भी कोशिका, विशेषकर वे जो युग्मकों के निर्माण के लिए उत्तरदायी हैं, जब आयनकारी $\mathrm{X}$-किरणन विकिरण के संपर्क में आती है तो यह अणु के भीतर मौजूद विभिन्न बंधों के टूटने को प्रेरित कर सकती है। यदि ऐसा टूटाव DNA के फॉस्फोडाइएस्टर बंध में होता है तो इससे उसमें कुछ न्यूक्लियोटाइडों की हानि हो सकती है। इससे एक या कुछ न्यूक्लियोटाइडों की विलोपन के कारण फ्रेम-शिफ्ट उत्परिवर्तन उत्पन्न हो सकता है। जीनों के रीडिंग फ्रेम में इस प्रकार के परिवर्तन का परिणाम समझा जा सकता है क्योंकि यह गलत RNA प्रतिलेखन और जीन की अभिव्यक्ति न होना या परिवर्तित अभिव्यक्ति की ओर ले जा सकता है। यहाँ तक कि अनायनकारी विकिरण जैसे UV किरणें भी DNA अणु के भीतर इलेक्ट्रॉन के उत्साहन का कारण बन सकती हैं। ये उत्साहित अणु अधिक क्रियाशील हो सकते हैं और विलोपन या प्रतिस्थापन प्रकार के उत्परिवर्तन को प्रेरित कर सकते हैं।
इसी प्रकार, बेस एनालॉग जैसे 5-ब्रोमोयूरेसिल जब अपने कीटो रूप में उपस्थित होता है तो यह DNA में एडेनिन के विरुद्ध पूरक बेस के रूप में वरीयता से समाविष्ट हो सकता है। हालाँकि, यदि यह अपने टॉटोमेरिक एनॉल रूप में रूपांतरित हो जाता है तो 5-ब्रोमोयूरेसिल ग्वानिन न्यूक्लियोटाइड के साथ युग्मन कर सकता है जिससे $A=T$ युग्म का $G \equiv C$ युग्म में प्रतिस्थापन हो जाता है (चित्र 7.34)।
एल्किलेटिंग एजेंट जैसे EMS डीएनए न्यूक्लियोटाइड को या तो 7वें स्थान पर नाइट्रोजन पर या 6वें स्थान पर ऑक्सीजन पर एथिलेट करता है। ऐसा एल्किलेशन युग्मन गुण को बदल देता है। उदाहरण के लिए, एथिलेटेड ग्वानिन न्यूक्लियोटाइड थाइमिन के साथ युग्मन बनाता है, जिसका अर्थ है कि डीएनए में $G \equiv C$ युग्म $A=T$ युग्म में उत्परिवर्तित हो जाएगा। कई ऐसे रासायनिक उत्परिवर्तजन युग्मन गुण में परिवर्तन की प्रक्रिया द्वारा उत्परिवर्तन उत्पन्न करते हैं।
7.8 डीएनए मरम्मत
उपरोक्त उत्परिवर्तन के विवरण से शायद यह समझ में आ सकता है कि उत्परिवर्तन की दर चाहे स्वतः हो या प्रेरित, बहुत अधिक होनी चाहिए। लेकिन ऐसी उच्च उत्परिवर्तन दर देखी नहीं जाती। साथ ही, इतनी उच्च उत्परिवर्तन दर की अपेक्षा भी नहीं की जाती, यह विचार करते हुए कि आनुवंशिक पदार्थ विश्वसनीय स्थिर तरीके से स्थानांतरित होता है। विभिन्न जीवों में आणविक प्रक्रियाओं की समझ से यह पता चला है कि ऐसी तंत्र मौजूद हैं जिनसे अधिकांश त्रुटियों को सुधारा भी जाता है। ऐसे कई तंत्र जीवाणु प्रणाली $E$. coli में पहचाने गए हैं। इनमें से कुछ को हम तंत्र को समझने के लिए देखेंगे।
हिंसात्मक मरम्मत- यह एक ऐसी प्रक्रिया है जिसमें DNA में बदले या संशोधित आधारों को क्रमबद्ध रूप से पहचानकर हटाया जाता है और फिर एंजाइमैटिक बाइंडिंग द्वारा उन्हें निकाल दिया जाता है। इस प्रकार बनाया गया अंतराल अंततः एक DNA पॉलिमरेज एंजाइम द्वारा भर दिया जाता है जिसके लिए अपरिवर्तित स्ट्रैंड को टेम्पलेट के रूप में उपयोग किया जाता है। एक ऐसी ही प्रक्रिया में DNA ग्लाइकोसिलेज नामक एंजाइम द्वारा बदले गए आधारों की पहचान की जाती है, जो विशेष रूप से या तो डीएमिनेटेड या ऑक्सीडाइज्ड आधारों को पहचानता है और फिर बदले गए आधार और डिऑक्सीराइबोज शर्करा के बीच बंध को काटता है। परिणामस्वरूप, DNA के एक स्ट्रैंड में एक ऐसा स्थान बनता है जिसमें कोई न्यूक्लियोटाइड नहीं होता, जिसे AP साइट कहा जाता है (जो या तो अप्यूरिनिक या अपायरीमिडिनिक साइट के लिए होता है)। एक विशिष्ट एंजाइम AP एंडोन्यूक्लिएस इस AP साइट को पहचानता है और शर्कराफॉस्फेट समूह को हटाकर उस स्थान पर एक अंतराल बना देता है। अंत में, एक DNA पॉलिमरेज सही न्यूक्लियोटाइड को पूरक स्ट्रैंड के अनुसार रखकर अंतराल को भरता है और फिर DNA लाइगेज एंजाइम द्वारा निक को जोड़ता है (चित्र 7.35)। इस प्रकार की हिंसात्मक मरम्मत को आधार हिंसात्मक मरम्मत भी कहा जाता है।
अन्य कटिंग-मरम्मत (excision repair) तंत्र भी मौजूद हैं जिनमें डीएनए के तुलनात्मक रूप से बड़े बदले हुए हिस्सों की मरम्मत हो सकती है। एक ऐसा उदाहरण थाइमीन डाइमर (thymine dimer) द्वारा बना क्षति की मरम्मत है। ऐसा डाइमर दो संलग्न थाइमीन न्यूक्लियोटाइडों के कार्बनों के बीच पराबैंगनी किरणों के कारण हुई फोटोरासायनिक प्रतिक्रिया के फलस्वरूप एक सहसंयोजक बंध के रूप में बन सकता है। इस तरह के डाइमर बनने के परिणामों को इस तथ्य से आसानी से समझा जा सकता है कि यह अगले प्रतिकृतिक चक्र (replication cycle) के दौरान डाइमर किसी भी न्यूक्लियोटाइड के साथ युग्मित नहीं हो पाता, जिससे विलोपन प्रकार का फ्रेमशिफ्ट उत्परिवर्तन (frameshift mutation) उत्पन्न हो सकता है। इस प्रकार की क्षति के लिए न्यूक्लियोटाइड कटिंग-मरम्मत (nucleotide excision repair) नामक तंत्र थोड़ा जटिल है, जिसमें एक विशिष्ट त्रिमात्रिक (trimeric) प्रोटीन जिसे Uvr (Uvr का अर्थ है अल्ट्रा वायलेट मरम्मत) प्रोटीन कहा जाता है, डाइमर स्थल को पहचानता है, उससे बंधता है और उसे मोड़ता है। त्रिमात्रिक प्रोटीन की UvrA पॉलीपेप्टाइड की दो इकाइयाँ स्थल को छोड़ देती हैं और एक अन्य प्रोटीन UvrB क्षतिग्रस्त स्थल पर डीएनए अणु के साथ एक संकुल (complex) बनाता है और 3’ छोर की ओर 5वें फॉस्फोडाइएस्टर बंध को तोड़ता है। एक अन्य प्रोटीन UvrC भी क्षतिग्रस्त स्थल पर कार्य करता है और 5’ छोर की ओर 8वें फॉस्फोडाइएस्टर बंध को तोड़ता है। इस प्रकार क्षतिग्रस्त डाइमर वाली डीएनए स्ट्रैंड से 12 न्यूक्लियोटाइडों का एक हिस्सा काट लिया जाता है। इस 12 न्यूक्लियोटाइड लंबे अंतराल को डीएनए पॉलिमरेज़ I द्वारा भरा जाता है और तत्पश्चात डीएनए लाइगेज द्वारा निक (nick) को सील किया जाता है (चित्र 7.36)।
अनुवाद (हिन्दी):
बेमेल मरम्मत (Mismatch repair)
कभी-कभी DNA प्रतिकृतिकरण (replication) के दौरान गलत न्यूक्लियोटाइड जुड़ जाता है। इस गलती को सुधारने के लिए कोशिका में एक विशेष तंत्र मौजूद है, जिसमें चार प्रकार के प्रोटीन—MutH, MutL, MutS और MutT—काम करते हैं। यह मरम्मत प्रक्रिया प्रतिकृतिकरण की “प्रूफ-रीडिंग” जैसी भी कार्य करती है।
MutS प्रोटीन सबसे पहले बेमेल (mismatch) को पहचानता है। इसके बाद MutH और MutL प्रोटीन भी जुड़कर एक संकुल (complex) बनाते हैं। MutH की विशिष्ट एंडोन्यूक्लिएस गतिविधि DNA की एक श्रृंखला को 5′ या 3′ दिशा में काट देती है; यह कट 1000 या उससे अधिक न्यूक्लियोटाइड दूर हो सकता है। इसके बाद बेमेल युक्त भाग को बाहर निकाल दिया जाता है। इस प्रकार बने रिक्त स्थान (gap) को DNA पॉलिमरेज़ भरता है और अंत में DNA लाइगेज़ द्वारा निक को जोड़ दिया जाता है।
कुछ अन्य डीएनए मरम्मत तंत्रों को भी प्रोकैरियोट्स और यूकैरियोट्स दोनों में देखा और अध्ययन किया गया है। आप उन्हें अपने उच्च स्तर की कक्षाओं में पढ़ेंगे। हालांकि, उत्परिवर्तन और मरम्मत तंत्र के विवरण के आधार पर जेनेटिक सूचना की स्थिर प्रकृति को इसकी परिवर्तनशीलता की क्षमता के सापेक्ष सराहा जा सकता है।
7.9 पुनःसंयोजन
पुनःसंयोजन की अवधारणा को जेनेटिसिस्टों ने सन् 1900 में मेंडल के वंशागति के सिद्धांतों की पुनःखोज के तुरंत बाद देखा। स्वीट पी में डब्ल्यू. बेटेसन और आर. सी. पन्नेट द्वारा किए गए प्रयोगों ने स्पष्ट रूप से स्थापित किया कि सभी जीन स्वतंत्र रूप से वितरित नहीं होते। यह स्वीट पी में किए गए द्विगुणसूत्रीय संकरण में स्पष्ट था। लाल फूल और लंबे परागकणों वाली शुद्ध पंक्ति को सफेद फूल और छोटे परागकणों वाली पंक्ति से संकरित करने पर अपेक्षित लाल फूल और लंबे परागकणों वाली संतति प्राप्त हुई। लेकिन स्व-संकरित $\mathrm{F}_{2}$ संतति में 50% से अधिक संतति में फूल के रंग और परागकण के आकार के लिए माता-पिता के संयोजन लाल-लंबे और सफेद-छोटे थे। पुनःसंयोजक, अर्थात् छोटे परागकणों वाले लाल फूल और लंबे परागकणों वाले सफेद फूल 50% से कम थे। यह अपेक्षित 9:3:3:1 अनुपात से विचलन था (चित्र 6.8)।
हालांकि, बेटसन और पुनेट द्वारा प्रस्तुत प्रारंभिक व्याख्या भिन्न थी, परंतु बाद में इसे लिंकेज की घटना के रूप में स्थापित किया गया, जिसमें एक ही गुणसूत्र पर स्थित जीन एक साथ वंशानुगत होते हैं। पुनः संयोजकों की उपस्थिति को क्रॉसिंग ओवर की घटना से जोड़ा गया, जिसमें मीओसिस के दौरान समजातीय गुणसूत्रों के भागों का आदान-प्रदान पुनः संयोजकों को जन्म दे सकता है। स्पष्ट है कि ऐसे पुनः संयोजकों का प्रतिशत स्वतंत्र रूप से वर्गीकृत लक्षणों की तुलना में कम होने की अपेक्षा है, जैसा कि पहले वर्णित प्रयोग में देखा गया है। बाद में, थॉमस हंट मॉर्गन द्वारा ड्रोसोफिला मेलानोगास्टर में किए गए समान प्रयोगों ने लिंकेज और पुनः संयोजन के कई उदाहरण प्रदान किए। केवल इतना ही नहीं, पुनः संयोजकों की आवृत्ति या प्रतिशत के आधार पर कई जीवों के गुणसूत्रों का भौतिक मानचित्र तैयार किया गया। ऐसे गुणसूत्र के भौतिक मानचित्र को तैयार करते समय दो लक्षणों के बीच $1 %$ पुनः संयोजकों को 1 मानचित्र इकाई या 1 सेंटीमॉर्गन (cM) की दूरी पर निर्दिष्ट किया गया।
इसके अतिरिक्त, मियोसिस के साइटोलॉजिकल अध्ययनों और प्रथम मियोटिक विभाजन में कायस्मेटा की उपस्थिति, जब समजात गुणसूत्र युग्म अलग होते हैं (अपचायी विभाजन), ने पुनःसंयोजकों और समजात गुणसूत्र युग्म के भागों के आदान-प्रदान के बीच प्रत्यक्ष संबंध को समझने में अंतर्दृष्टि प्रदान की। पहले प्रमाणों में से एक, जिसने दर्शाया कि क्रॉसिंग ओवर पुनःसंयोजन के लिए उत्तरदायी है, वह मक्का में हेरिएट क्रेइंगटन और बारबरा मैक्लिनटॉक (1931) द्वारा किया गया शास्त्रीय प्रयोग था। अपने सावधानीपूर्ण अवलोकन के आधार पर उन्होंने पाया कि कुछ समजात गुणसूत्र आकृति-विज्ञानतः भिन्न हो सकते हैं। उनकी जांच में, गुणसूत्र 9 के दो प्रकार पहचाने गए। युग्म में से एक सामान्य था जबकि दूसरे के एक सिरे पर विषमरंजक गांठ (heterochromatic knob) और दूसरे सिरे पर दूसरे गुणसूत्र का एक छोटा टुकड़ा था। आपने पहले ही गुणसूत्रीय विसंगति का अध्ययन किया है और समझ गए हैं कि ये कैसे उत्पन्न हो सकती हैं। उनके प्रयोग में, पुनःसंयोजन को समझने के लिए दो लक्षणों को मार्कर के रूप में प्रयोग किया गया। एक वह जीन था जो बीज का रंग निर्धारित करता है, अर्थात् रंगीन (C) बनाम बिना रंग के (c)। दूसरा वह जीन था जो बीज की बनावट निर्धारित करता है, अर्थात् मांडयुक्त (Wx) बनाम मोमी (wx)। चित्र 7.37 के अनुसार क्रॉस करने पर यह प्रमाण मिलता है कि क्रॉसिंग ओवर के दौरान समजात गुणसूत्रों के भागों का आदान-प्रदान होता है
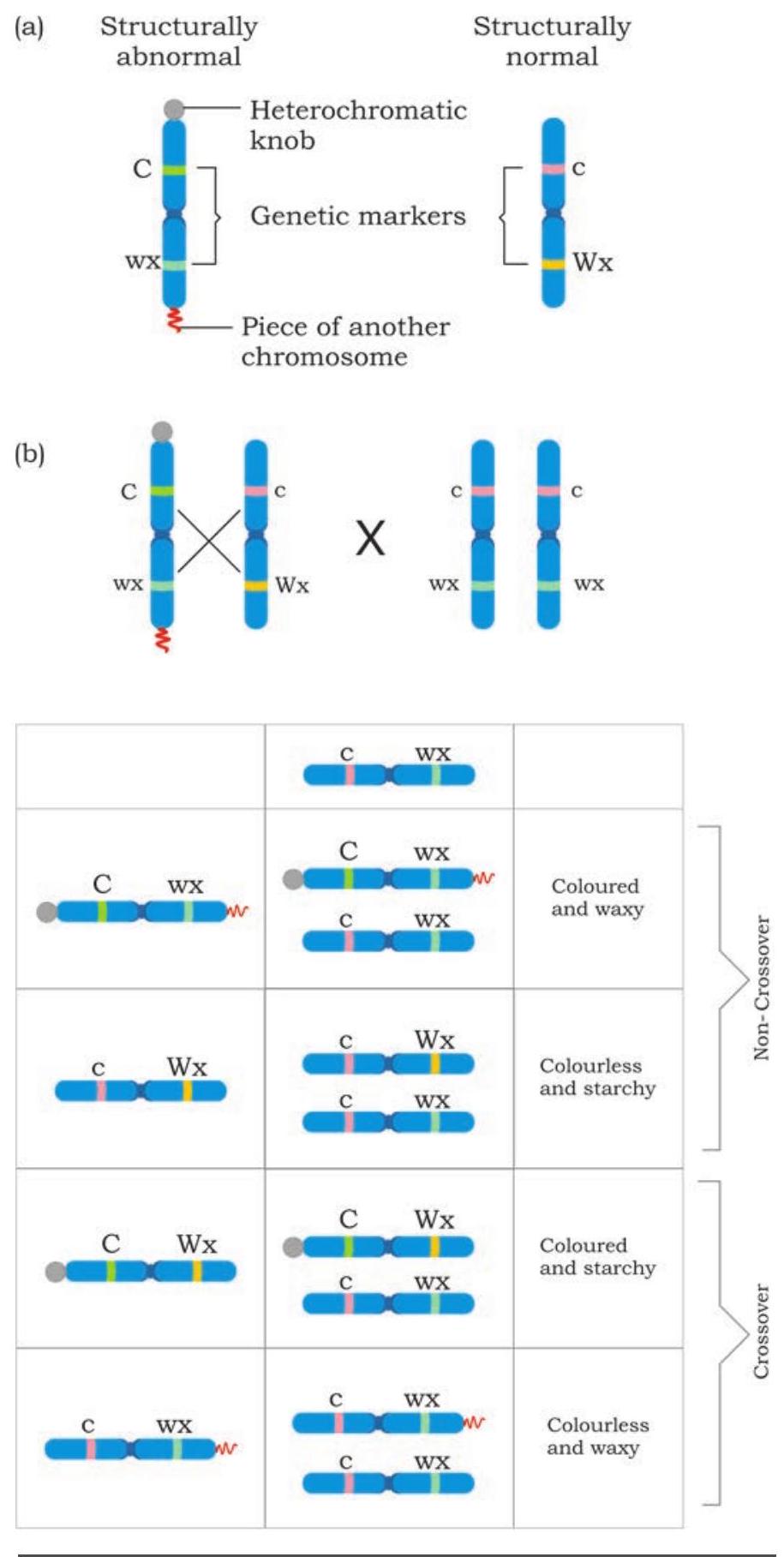
चित्र 7.37: क्रॉसिंग ओवर का प्रायोगिक प्रमाण (a) एक गुणसूत्र पर कोशिकीय चिह्न और दूसरा सामान्य गुणसूत्र (b) टेस्ट क्रॉस का परिणाम जिसमें नॉन-क्रॉस ओवर और क्रॉस ओवर संतति दिखाई गई है। जब पुनःसंयोजी संतानों की जांच गुणसूत्र के आदान-प्रदान हुए भाग के लिए की गई, तो पाया गया कि पुनःसंयोजी, अर्थात् या तो रंगीन स्टार्ची बीज वाले या रंगहीन वैक्सी बीज वाले, के गुणसूत्र 9 के भिन्न आकृति-विज्ञानी रूप थे। यहाँ पुनःसंयोजियों में केवल एक विचित्र चिह्न कोशिकीय रूप से पाया गया, जबकि दोनों चिह्नों की उपस्थिति एक ही गुणसूत्र पर होती है, जिससे यह तथ्य स्थापित होता है कि पुनःसंयोजन के दौरान समजात गुणसूत्रों के भागों का आदान-प्रदान होता है।
7.10 जीन अभिव्यक्ति का नियमन
क्या आप जानते हैं कि बहुकोशिकीय जीव में संरचना और कार्य में भिन्न कई प्रकार की कोशिकाएँ होती हैं, फिर भी उनके जीन समान होते हैं? ऐसा इसलिए है क्योंकि सभी कोशिकाएँ युग्मनज (zygote) से समितोटिक विभाजन द्वारा उत्पन्न होती हैं। किसी जीव की सभी क्रियाएँ जीनों द्वारा नियंत्रित होती हैं। जीव के अधिकांश जीन प्रोटीन बनाकर अपना अभिव्यक्तन करते हैं। सभी जीन सभी कोशिकाओं में अभिव्यक्त नहीं होते क्योंकि उनके उत्पाद एक समय पर आवश्यक नहीं होते। केवल वे जीन ही अभिव्यक्त होते हैं जिनके उत्पाद उस कोशिका में आवश्यक होते हैं, जबकि अन्य जीन अभिव्यक्त नहीं होते क्योंकि उनके उत्पाद उस समय कोशिका द्वारा आवश्यक नहीं होते। जीन क्रिया का यह ‘चालू और बंद’ तंत्र जीन अभिव्यक्तन का नियमन या जीन क्रिया का नियमन कहलाता है।
निम्न स्तर के जीव जैसे जीवाणु विभिन्न पर्यावरणीय परिस्थितियों का सामना करते हैं। उदाहरण के लिए, ई. कोलाई (E. coli) हमारे बड़े आंत में रहता है। हमारी खाने की आदतें इस जीवाणु के लिए उपलब्ध पोषक तत्वों को निर्धारित करती हैं। जब ग्लूकोस उपलब्ध होता है, तो वे जीन अभिव्यक्त होते हैं जिनके उत्पाद (एंजाइम) ऊर्जा उत्पन्न करने के लिए उसे तोड़ते हैं। यदि लैक्टोस या कोई अन्य शर्करा उपलब्ध हो तो कुछ अन्य जीन अभिव्यक्त होते हैं जिनके उत्पाद उसे तोड़कर ऊर्जा उत्पन्न करते हैं। यह दर्शाता है कि कोशिका की आवश्यकता के अनुसार विशिष्ट समय पर विशिष्ट जीन अभिव्यक्त होते हैं।
जीनों की अभिव्यक्ति को जीनोटाइप से फ़ीनोटाइप तक सूचना के प्रवाह के मार्ग के विभिन्न चरणों पर नियंत्रित किया जा सकता है। नियंत्रण क्रोमेटिन स्तर, ट्रांसक्रिप्शन स्तर, mRNA प्रोसेसिंग (यूकैरियोट्स), mRNA के परिवहन और ट्रांसलेशन स्तर पर हो सकता है (चित्र 7.38)। प्रोकैरियोटिक और यूकैरियोटिक दोनों प्रणालियों में ट्रांसक्रिप्शन प्रारंभ जीन नियंत्रण का एक महत्वपूर्ण बिंदु है क्योंकि कोशिका न केवल यह निर्णय लेती है कि कौन-सा जीन व्यक्त होना है, बल्कि उसकी अभिव्यक्ति की मात्रा भी। किसी प्रजाति या जीव के अधिकांश कोशिकाओं में कुछ जीन लगभग स्थिर स्तर पर व्यक्त होते हैं और इन्हें ‘हाउसकीपिंग जीन’ या ‘स्थायी जीन’ कहा जाता है। इन जीनों के उत्पाद हर समय आवश्यक होते हैं। सिट्रिक अम्ल चक्र जैसे केंद्रीय उपापचयी मार्गों के चरणों को उत्प्रेरित करने वाले एंजाइमों को कोडित करने वाले जीन इस श्रेणी में आते हैं। जीनों की इस प्रकार की अनियंत्रित अभिव्यक्ति को स्थायी जीन
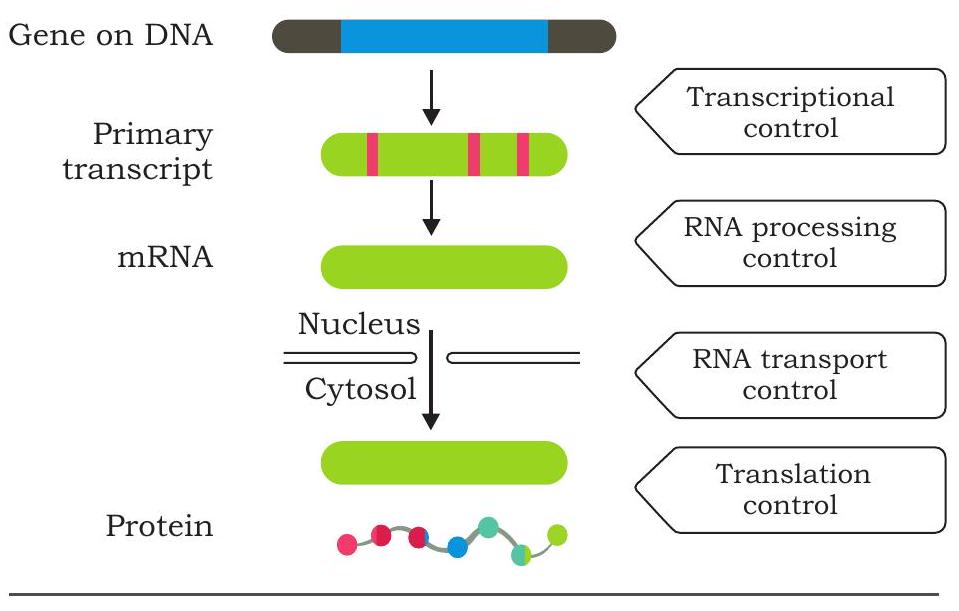
चित्र 7.38 जीन अभिव्यक्ति के नियंत्रण के स्तर अभिव्यक्ति कहा जाता है। परंतु अधिकांश जीनों की अभिव्यक्ति की दर कोशिकाओं में प्राप्त आण्विक संकेतों के अनुसार बदलती रहती है। इन जीनों के उत्पाद का स्तर कोशिका की आवश्यकता के अनुसार बढ़ता और घटता रहता है। इस प्रकार के नियंत्रण को विनियमित जीन अभिव्यक्ति कहा जाता है।
7.10.1 जीवाणुओं में जीन अभिव्यक्ति का नियंत्रण
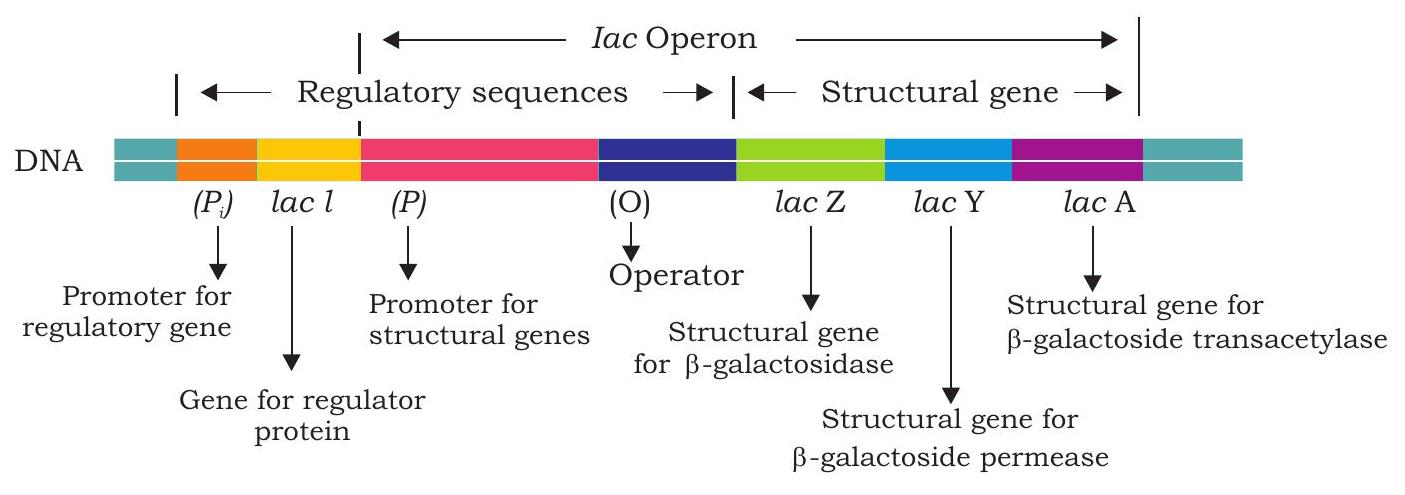
जीन अभिव्यक्ति के नियमन की क्रिया प्रथमतः जीवाणु कोशिकाओं में अध्ययन की गई। जीवाणुओं में क्रियात्मक रूप से सम्बद्ध जीनों की संरचना यूकैरियोट्स से भिन्न होती है। जीवाणुओं में, जो जीन सम्बन्धित कार्य करते हैं वे एक समूह में समूहीकृत होते हैं और प्रायः एक साथ एकल mRNA अणु में ट्रांसक्राइब होते हैं। सम्बन्धित जीनों को समूहीकृत करने का लाभ यह है कि समूह के सभी जीनों को एक ही ‘ऑन-ऑफ स्विच’ नियंत्रित कर सकता है। इसका अर्थ है कि समूह के सभी जीन समन्वित रूप से नियंत्रित होते हैं। दूसरी ओर, यूकैरियोट का प्रत्येक जीन एक पृथक mRNA में ट्रांसक्राइब होता है। समूहीकृत संरचनात्मक जीनों का एक समूह जो एक साथ प्रमोटर और अतिरिक्त नियंत्रण अनुक्रमों (जो ट्रांसक्रिप्शन को नियंत्रित करते हैं) के साथ ट्रांसक्राइब होता है, को ‘ऑपरॉन’ कहा जाता है।
एक प्रतिनिधि ऑपरॉन (चित्र 7.39) के एक सिरे पर संरचनात्मक जीनों या सिस्ट्रॉन्स का एक समुच्चय होता है (जो चयापचय में संलग्न प्रोटीनों को कोडित करते हैं) जो ट्रांसक्राइब होते हैं और फिर अनुवादित होकर विभिन्न प्रोटीन उत्पन्न करते हैं। प्रथम संरचनात्मक जीन के अपस्ट्रीम ‘प्रमोटर’ होता है जो संरचनात्मक जीनों के ट्रांसक्रिप्शन को नियंत्रित करता है। RNA पॉलिमरेज बंधन स्थल प्रमोटर में स्थित होता है। एक DNA खंड जिसे ‘ऑपरेटर’ कहा जाता है, प्रमोटर के भीतर या प्रमोटर और प्रथम संरचनात्मक जीन के बीच स्थित होता है, जो RNA पॉलिमरेज की जीनों तक पहुँच को नियंत्रित करता है।
चित्र 7.39: एक ऑपरॉन की संरचना
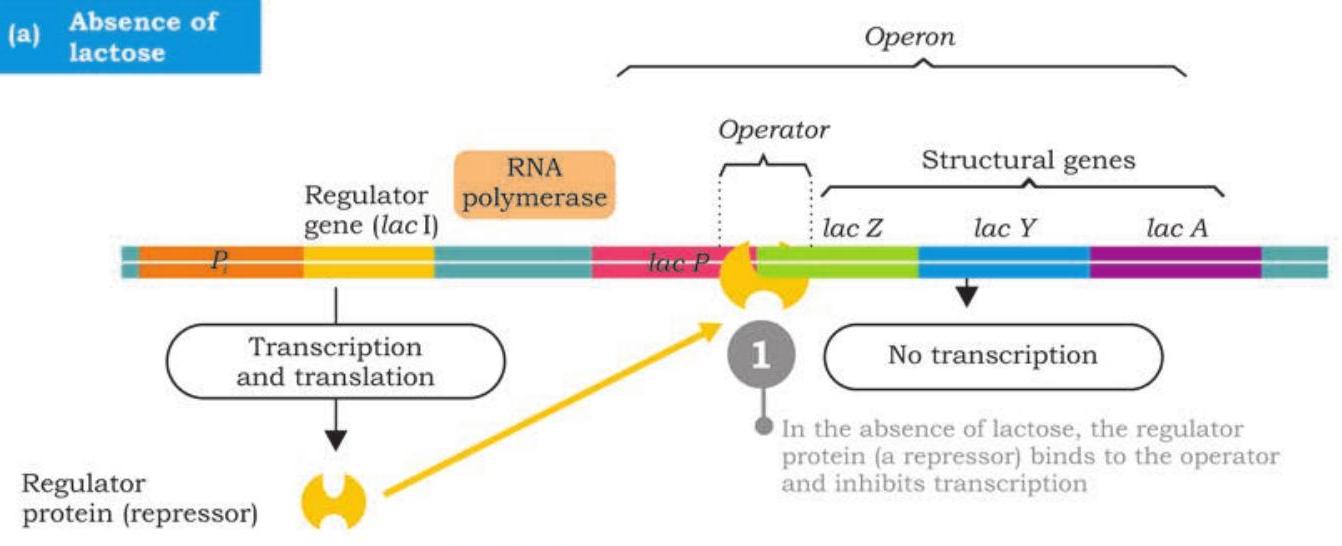
ऑपरेटर उस स्थल को कहते हैं जहाँ रिप्रेसर प्रोटीन बँधता है; रिप्रेसर ऑपरेटर से जुड़कर ‘ऑपरेटर-रिप्रेसर’ समिश्र बनाता है। जब रिप्रेसर ऑपरेटर से बँधा होता है, तो संरचनात्मक जीनों की प्रतिलेखन-प्रक्रिया नहीं चल पाती। ऑपरॉन के प्रमोटर से ऊपर की ओर एक ‘नियामक जीन’ (regulator gene) स्थित होता है। यद्यपि यह जीन स्वयं ऑपरॉन का भाग नहीं माना जाता, फिर भी वह संरचनात्मक जीनों के प्रतिलेखन को नियंत्रित करता है। इसका अपना प्रमोटर होता है और यह एक छोटी mRNA बनाता है, जिसका अनुवाद होकर एक नियामक प्रोटीन (रिप्रेसर) तैयार होता है। रिप्रेसर या तो सक्रिय (active) हो सकता है या निष्क्रिय (inactive)। सक्रिय रिप्रेसर प्रोटीन ऑपरॉन के ऑपरेटर से बँधकर RNA पॉलिमरेज को प्रमोटर से जुड़ने से रोकता है, जिससे संरचनात्मक जीनों का प्रतिलेखन बाधित हो जाता है।
ओपेरॉन के नियमन की क्रिया-विधि का वर्णन पहली बार फ्रांस्वा जैकब और जैक मोनोड ने 1961 में किया था और उन्होंने ई. कोलाई कोशिकाओं में लैक्टोज चयापचय के आनुवंशिक नियंत्रण के लिए ‘ओपेरॉन मॉडल’ प्रस्तावित किया। ओपेरॉन के दो प्रकार होते हैं, प्रेरणीय ओपेरॉन और दमनकारी ओपेरॉन, जो प्रभावक अणु के प्रति अपनी प्रतिक्रिया के स्वभाव के आधार पर वर्गीकृत किए जाते हैं। प्रेरणीय ओपेरॉनों के मामले में, प्रभावक अणुओं को प्रेरक (उपस्ट्रेट) कहा जाता है, जब ये उपस्थित होते हैं तो सक्रिय दमनकारी से बांधते हैं और उसे निष्क्रिय कर देते हैं। निष्क्रिय दमनकारी-प्रेरक संकुल ऑपरेटर से नहीं बंध सकता, और ओपेरॉन में संरचनात्मक जीनों का अनुलेखन चालू (प्रेरित) हो जाता है और तत्पश्चात प्रोटीन (एंजाइम) का अनुवाद होता है। वे एंजाइम जिनके उत्पादन में वृद्धि उस उपस्ट्रेट की उपस्थिति से हो सकती है जिस पर वे कार्य करते हैं, प्रेरणीय एंजाइम कहलाते हैं और ऐसे एंजाइम के संश्लेषण के लिए उत्तरदायी आनुवंशिक तंत्र को प्रेरणीय तंत्र कहा जाता है। दमनकारी ओपेरॉनों के मामले में, प्रभावक अणुओं को सह-दमनकारी (अंत-उत्पाद) कहा जाता है। जब सह-दमनकारी निष्क्रिय दमनकारियों से बांधते हैं, तो दमनकारी-सह-दमनकारी संकुल सक्रिय हो जाता है, ऑपरेटर से बांधता है और आरएनए पॉलिमरेज को संरचनात्मक जीनों का अनुलेखन करने से रोकता है। उदाहरण के लिए, जब बाहर से कोई अमीनो अम्ल नहीं दिया जाता, तो ई. कोलाई कोशिकाएं विभिन्न अमीनो अम्लों के संश्लेषण के लिए आवश्यक सभी एंजाइमों का संश्लेषण कर सकती हैं। यदि कोई विशिष्ट अमीनो अम्ल, उदाहरण के लिए, हिस्टिडिन, डाला जाता है, तो हिस्टिडिन संश्लेषित करने वाले एंजाइम का उत्पादन घट जाता है। ऐसे तंत्र में, अंत-उत्पाद की वृद्धि से जैवसंश्लेषण के लिए आवश्यक एंजाइमों के संश्लेषण की जांच हो जाती है। ऐसे एंजाइम जिनके संश्लेषण की जांच अंत-उत्पाद की वृद्धि से हो सकती है, दमनकारी एंजाइम कहलाते हैं और ऐसे आनुवंशिक तंत्र को दमनकारी तंत्र कहा जाता है। अनुलेखन नियंत्रण के दो प्रकार होते हैं: नकारात्मक और सकारात्मक नियंत्रण। नकारात्मक नियंत्रण में, नियामक प्रोटीन एक दमनकारी होता है जो अनुलेखन को रोकता है। सकारात्मक नियंत्रण में, नियामक प्रोटीन एक सक्रियक होता है जो अनुलेखन को उत्तेजित करता है।
7.10.2 लैक ऑपरॉन - एक प्रेरणीय ऑपरॉन
दूध में पाया जाने वाला लैक्टोज़ (दुग्ध शर्करा, एक डाइसैकेराइड) जो कि एक $\beta$-गैलेक्टोसाइड है, जब कोई व्यक्ति दूध पीता है तो यह बैक्टीरिया $E$. coli के लिए बृहदान्त्र में उपलब्ध होता है। बैक्टीरिया लैक्टोज़ का उपयोग ऊर्जा के साथ-साथ कार्बन के स्रोत के रूप में करता है जब यह एंजाइम $\beta$-गैलेक्टोसिडेस द्वारा ग्लूकोज़ और गैलेक्टोज़ में टूट जाता है।
चित्र 7.40: लैक ऑपरॉन की संरचना
लैक ऑपरॉन
जब $E$. coli लैक्टोज़ की अनुपस्थिति में वृद्धि कर रहा होता है, तो कोशिकाओं में $\beta$-गैलेक्टोसिडेस की बहुत कम मात्रा होती है, लेकिन जब बैक्टीरिया के वातावरण में लैक्टोज़ मिलाया जाता है, तो कोशिकाओं में $\beta$-गैलेक्टोसिडेस अणुओं की संख्या 2 से 3 मिनट के भीतर कई गुना बढ़ जाती है।
लैक ऑपरॉन में तीन संरचनात्मक जीन होते हैं; lacZ, lacY और lacA जो तीन अलग-अलग प्रोटीनों को कोडित करते हैं (चित्र 7.40)। lac $Z$ जीन $\boldsymbol{\beta}$-गैलेक्टोसिडेस को कोडित करता है जो लैक्टोज़ को ग्लूकोज़ और गैलेक्टोज़ में तोड़ता है। यह एंजाइम लैक्टोज़ को एलोलैक्टोज़ में भी बदल सकता है जो लैक ऑपरॉन में एक प्रेरक के रूप में कार्य करता है। lac $Y$ जीन $\boldsymbol{\beta}$-गैलेक्टोसाइड परमीएस को कोडित करता है, जो एक झिल्ली प्रोटीन है जो लैक्टोज़ को सक्रिय रूप से कोशिका के अंदर पहुंचाता है। lacA जीन $\boldsymbol{\beta}$-गैलेक्टोसाइड ट्रांसएसिटिलेज को कोडित करता है, लेकिन लैक्टोज चयापचय में इसकी भूमिका अभी तक ज्ञात नहीं है।
रेगुलेटर प्रोटीन (रिप्रेसर) जीन (lacI)
चित्र 7.41: lac ऑपरॉन का नियमन
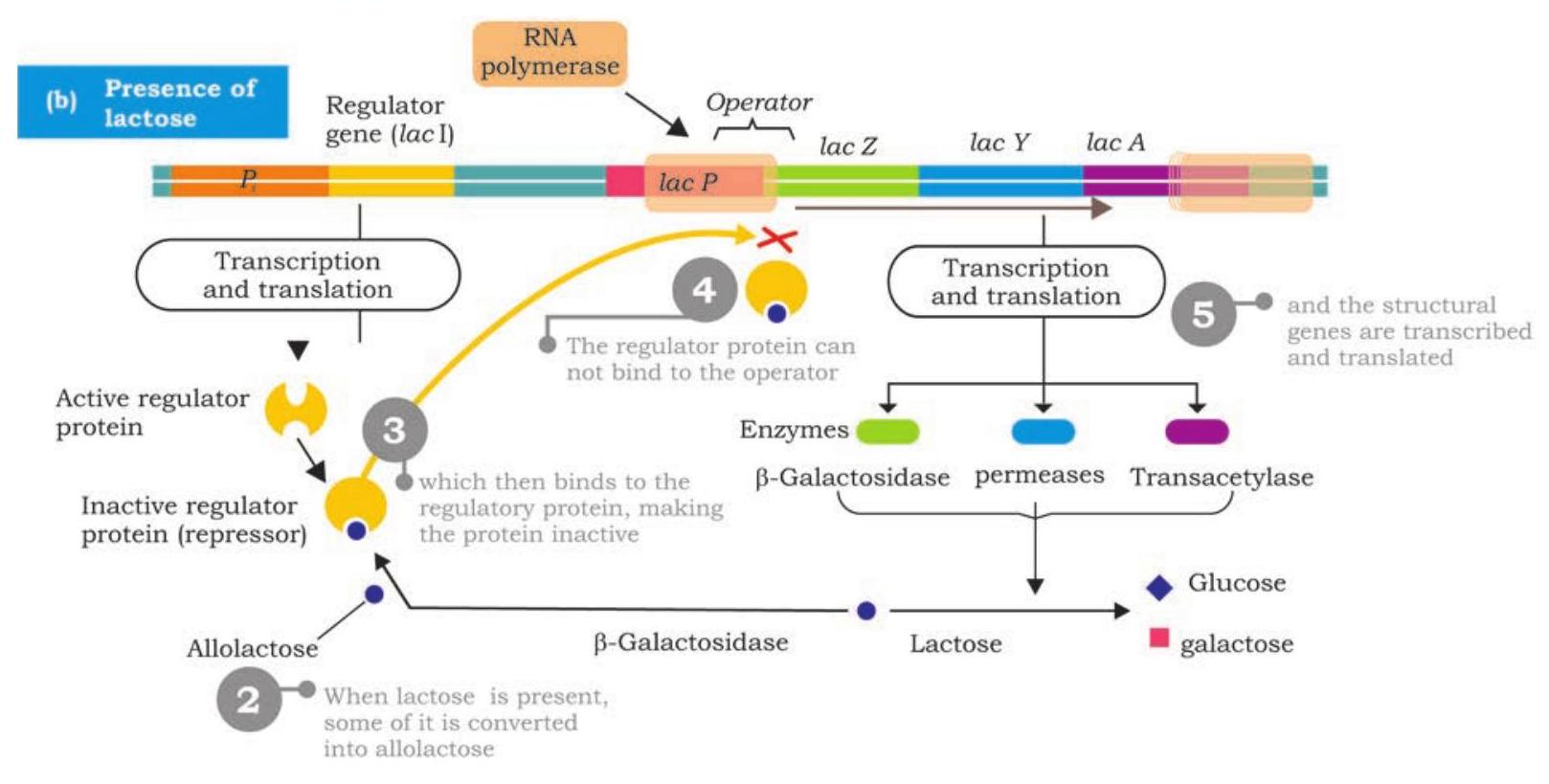
रेगुलेटर जीन जिसे lacI कहा जाता है, lac ऑपरॉन के प्रमोटर के ऊपरस्थित है और इसका अपना प्रमोटर होता है। इसे एक छोटे mRNA में ट्रांसक्राइब किया जाता है जिसे फिर एक रेगुलेटर प्रोटीन जिसे रिप्रेसर प्रोटीन कहा जाता है, में ट्रांसलेट किया जाता है। रिप्रेसर एक अलोस्टेरिक प्रोटीन है जिसमें दो बाइंडिंग साइट होते हैं; एक ऑपरॉन के ऑपरेटर से बाइंड करने के लिए और दूसरा इंड्यूसर (अलोलैक्टोज) से बाइंड करने के लिए।
जब E. coli कोशिकाओं में लैक्टोज़ उपस्थित नहीं होता है, तो lacI द्वारा कोडित रिप्रेसर प्रोटीन सक्रिय होता है और lac ऑपरेटर से बंधता है; यह भौतिक रूप से RNA पॉलिमरेज़ के बंधन को रोकता है और संरचनात्मक जीनों की ट्रांसक्रिप्शन को रोका जाता है (चित्र 7.41)। यह lac ऑपरॉन का नकारात्मक नियंत्रण है। जब तक रिप्रेसर ऑपरेटर से बंधा रहता है, कोई प्रोटीन नहीं बनता है। हालांकि, जब लैक्टोज़ उपस्थित होता है, तो $\beta$-गैलेक्टोसिडेज़ इसका कुछ भाग एलोलैक्टोज़ में परिवर्तित करता है। एलोलैक्टोज़ एक इंड्यूसर के रूप में कार्य करता है, सक्रिय रिप्रेसर से बंधता है और एक संरचनात्मक परिवर्तन का कारण बनता है जिससे वह निष्क्रिय हो जाता है। निष्क्रिय रिप्रेसर ऑपरेटर से बंधने में विफल रहता है और RNA पॉलिमरेज़ का बंधन अब अवरुद्ध नहीं होता है। RNA पॉलिमरेज़ अब lacZ, lacY और lacA को एक पॉलीसिस्ट्रॉनिक mRNA में ट्रांसक्राइब करता है जो लैक्टोज़ चयापचय के लिए आवश्यक तीन अलग-अलग एंजाइमों में अनुवादित होता है (चित्र 7.42)। लैक्टोज़ को तोड़ने वाले एंजाइमों का उत्पादन तब तक जारी रहता है जब तक पर्याप्त लैक्टोज़ अणु टूट नहीं जाते और फिर रिप्रेसर ऑपरेटर के साथ पुनः संयुक्त होकर एंजाइमों के उत्पादन को रोक देते हैं। lac ऑपरॉन एक इंड्यूसिबल ऑपरॉन है क्योंकि लैक्टोज़ की उपस्थिति $\beta$-गैलेक्टोसिडेज़, $\beta$-गैलेक्टोसाइड पर्मिएस और $\beta$-गैलेक्टोसाइड ट्रांसएसिटिलेज़ के उत्पादन को प्रेरित करती है।
lac ऑपरॉन के सकारात्मक नियंत्रण में, नियामक प्रोटीन, अर्थात् एक एक्टिवेटर, ऑपरेटर के अतिरिक्त किसी अन्य स्थान पर DNA से बंधता है। एक्टिवेटर उत्पन्न किया जाता है
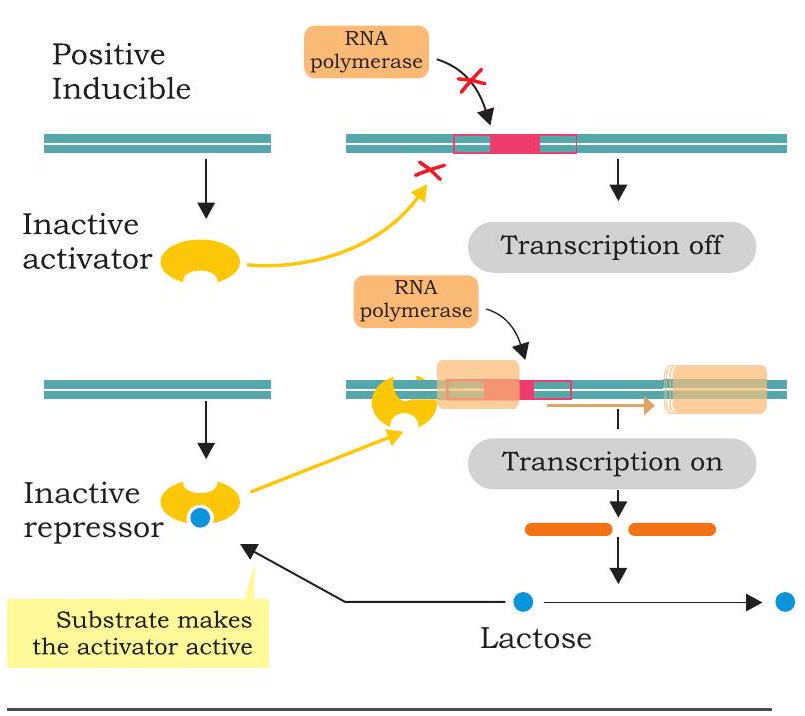
चित्र 7.42: लैक ऑपरॉन का सकारात्मक नियंत्रण निष्क्रिय अवस्था में है और डीएनए से बंधने में विफल रहता है (चित्र 7.42)। आरएनए पॉलिमरेज प्रमोटर से नहीं बंधता और ट्रांसक्रिप्शन बंद हो जाता है। जब इंड्यूसर निष्क्रिय एक्टिवेटर से जुड़कर उसे सक्रिय बनाता है, तो आरएनए पॉलिमरेज प्रमोटर से बंधता है और ट्रांसक्रिप्शन प्रारंभ करता है।
सारांश
- डीएनए को पहली बार 1869 में जोहान फ्रेडरिक मिशर द्वारा पस कोशिकाओं के केंद्रक से अलग किया गया था।
- एक कोशिका से दूसरी कोशिका में आनुवंशिक पदार्थ के स्थानांतरण की घटना, जो प्राप्तकर्ता कोशिका की आनुवंशिक संरचना को बदल देती है, रूपांतरण कहलाती है और इसकी खोज फ्रेडरिक ग्रिफिथ ने 1928 में की थी।
- ओसवाल्ड एवरी, कॉलिन मैकलियोड और मैकलिन मैककार्टी द्वारा किए गए प्रयोगों ने डीएनए को संभावित रूपांतरण कारक के रूप में उजागर किया।
- हर्शी और चेस द्वारा 1952 में किए गए प्रयोगों ने यह प्रबल प्रमाण दिया कि डीएनए आनुवंशिक पदार्थ है।
- जीन वंशानुक्रम की वह इकाई है जो किसी विशिष्ट लक्षण या चरित्र को नियंत्रित करती है और इसे वैकल्पिक रूपों में भी व्यक्त किया जा सकता है जिन्हें एलील कहा जाता है।
- आरएनए संश्लेषण के माध्यम से पॉलीपेप्टाइड श्रृंखला के संश्लेषण के जरिए डीएनए की अभिव्यक्ति आनुवंशिकी के केंद्रीय सिद्धांत को दर्शाती है।
- यह तथ्य कि एक जीन एक पॉलीपेप्टाइड को एन्कोड करता है, केंद्रीय सिद्धांत को भी ‘एक जीन, एक प्रोटीन’ से संशोधित कर ‘एक जीन, एक पॉलीपेप्टाइड’ कर दिया गया।
- प्रत्येक जीन को कार्य की इकाई, पुनर्संयोजन की इकाई और उत्परिवर्तन की इकाई होने की परीक्षा को पूरा करना चाहिए।
- वह प्रकार की डीएनए प्रतिकृति जिसमें माता-पिता की द्वैध डीएनए दो समान पुत्री द्वैध बनाती है, जिनमें से प्रत्येक एक माता-पिता वाली श्रृंखला और एक नवसंश्लेषित पुत्री श्रृंखला से बनी होती है, अर्ध-संरक्षी प्रतिकृति कहलाती है।
- यह मेसेलसन और स्टाहल थे जिन्होंने प्रतिकृति के बाद डीएनए की पुरानी और नई श्रृंखला को प्रयोगात्मक रूप से अलग किया, अपने प्रयोग में नाइट्रोजन के दो समस्थानिकों ${ }^{14} \mathrm{~N}$ और ${ }^{15} \mathrm{~N}$ का उपयोग करके।
- डीएनए की प्रतिकृति में प्रोकैरियोट्स और यूकैरियोट्स दोनों में कई एंजाइम और प्रोटीन शामिल होते हैं जैसे डीएनए पॉलिमरेज, प्राइमेज, हेलिकेज, सिंगल-स्ट्रैंड बाइंडिंग प्रोटीन, टोपोइसोमरेज, डीएनए लाइगेज, डीएनए-निर्भर आरएनए पॉलिमरेज।
- प्रतिकृति आमतौर पर डीएनए अनुक्रम पर एक विशिष्ट स्थान से शुरू होती है जिसे प्रतिकृति का उद्गम कहा जाता है।
- नई श्रृंखला जो लगातार $5^{\prime} \rightarrow 3^{\prime}$ दिशा में संश्लेषित होती है, अग्रणी श्रृंखला कहलाती है।
- ओकाजाकी खंडों के साथ असंतत रूप से संश्लेषित डीएनए श्रृंखलाओं को पिछड़ी श्रृंखला कहा जाता है।
- प्रतिलेखन एक प्रक्रिया है जिसके द्वारा आनुवंशिक सूचना डीएनए से मRNA में स्थानांतरित की जाती है।
- अनुवाद वह प्रक्रिया है जिसके द्वारा सूचना mRNA से पॉलीपेप्टाइड श्रृंखला में स्थानांतरित होती है और इसमें चार चरण होते हैं: (i) tRNA का आवेशन (ii) प्रारंभ (iii) विस्तार, और (iv) समापन।
- रिवर्स ट्रांसक्रिप्शन के मामले में, आनुवंशिक पदार्थ में मौजूद सूचना जो आरएनए है, पहले एकल श्रृंखला पूरक डीएनए श्रृंखला में स्थानांतरित की जाती है और फिर इसे द्विश्रृंखला डीएनए में बदल दिया जाता है।
- एक जीन के भीतर, सामान्य रूप से केवल एक ही न्यूक्लियोटाइड श्रृंखला को आरएनए में प्रतिलेखित किया जाता है। वह डीएनए श्रृंखला जिसकी न्यूक्लियोटाइड अनुक्रम mRNA के पूरक होती है, टेम्पलेट या एंटीसेंस श्रृंखला कहलाती है, जबकि दूसरी श्रृंखला जिसकी बेस अनुक्रम mRNA के समान होती है (DNA में T और RNA में U को छोड़कर), सेंस या कोडिंग श्रृंखला कहलाती है।
- प्रतिलेखन प्रक्रिया में प्रोकैरियोट्स और यूकैरियोट्स दोनों के लिए सामान्य प्रमुख चरणों में प्रारंभ, विस्तार और समापन शामिल हैं। इन चरणों के अतिरिक्त, यूकैरियोट्स में प्राथमिक प्रतिलेखन पश्च-प्रतिलेखन संशोधनों से गुजरते हैं जैसे कैपिंग, स्प्लाइसिंग, पॉली-एडेनिलेटेड पूंछ।
- आनुवंशिक कोड त्रिपल कोडॉन होते हैं, अर्थात् तीन बेसों के अद्वितीय संयोजन जो अपने संयोजनों के आधार पर किसी विशिष्ट अमीनो अम्ल के लिए कोड करते हैं। 64 कोडॉन होते हैं, जिनमें से 61 कोडॉन 20 अमीनो अम्लों के लिए कोड करते हैं।
- पॉलीराइबोसोम्स कई राइबोसोम्स से बने होते हैं जो एक ही mRNA से जुड़े होते हैं।
- उत्परिवर्तन आनुवंशिक पदार्थ में परिवर्तन है, अर्थात् डीएनए (कुछ वायरस में आरएनए) और इसे एक या कुछ न्यूक्लियोटाइड की जोड़, विलोपन और प्रतिस्थापन के रूप में वर्गीकृत किया जा सकता है।
- उत्परिवर्तन की उच्च दरें नहीं देखी जातीं क्योंकि डीएनए मरम्मत तंत्र मौजूद होता है जो एक्सीजन मरम्मत, मिसमैच मरम्मत आदि के माध्यम से हो सकता है।
- पुनर्संयोजन एक प्रक्रिया है जिसके दौरान समजात गुणसूत्रों के भागों का आदान-प्रदान होता है।
अभ्यास
1. जीन अभिव्यक्ति का क्या महत्व है? इसमें शामिल चरण कौन-कौन से हैं?
2. लैक ओपेरन के उदाहरण देकर प्रोकैरियोट्स में जीन अभ्यक्ति के नियमन की प्रक्रिया का वर्णन कीजिए।
3. DNA प्रतिकृतिकरण पर कोशिका से सभी प्रोटीनों के नष्ट हो जाने का क्या प्रभाव पड़ेगा?
4. UV किरणों से DNA की संरचना किस प्रकार प्रभावित होती है? इस प्रकार की विकिरण के कारण होने वाले उत्परिवर्तन के आणविक आधार और कोशिकाओं द्वारा उनके सुधार के लिए प्रयुक्त तंत्र की चर्चा कीजिए।
5. निम्नलिखित के बीच अंतर स्पष्ट कीजिए
(a) अग्रदार डोर और पश्चदार डोर
(b) प्रतिलेखन और अनुवाद
(c) संक्रमण और विपरिवर्तन उत्परिवर्तन
(d) कोडॉन और प्रतिकोडॉन
6. निम्नलिखित में से किस प्रकार की विकिरण कोशिकाओं के लिए सबसे कम हानिकारक होने की संभावना है?
(a) गामा किरणें
(b) पराबैंगनी किरणें
(c) $\mathrm{X}$ किरणें
(d) अल्फा किरणें
- निम्नलिखित में से किस DNA मरम्मत तंत्र में अपायरिमिडिनिक या अप्यूरिनिक (AP) स्थल बनता है?
(a) उच्छेदन मरम्मत
(b) असंगति मरम्मत
(c) उपर्युक्त दोनों
(d) उपर्युक्त में से कोई नहीं




