Chapter 05 Genome Technology and Engineering
जैवप्रौद्योगिकी और आण्विक जीवविज्ञान के क्षेत्र में क्रमिक विकास, विशेष रूप से जीनोम मानचित्रण और इसके प्रभाव में, ने अनुसंधान के लिए नए द्वार खोले हैं साथ ही इसके कई क्षेत्रों में अनुप्रयोगों को भी। जीनोम अनुसंधान में प्रगति ने एक ओर किसी भी जीव के ट्रांसक्रिप्टोम और प्रोटियोम को समझने की संभावना को बढ़ाया है, और दूसरी ओर जीनोमिक्स और संबंधित इंजीनियरिंग का रोगों की भविष्यवाणी और उपचार में उपयोग संभव हुआ है। फसल सुधार, जीन थेरेपी और पर्यावरणीय खतरों के निवारण में अनुप्रयोग ऐसे अन्य क्षेत्र हैं जिनमें जीनोम तकनीक की अत्यधिक क्षमता और गुंजाइश है। इस अध्याय में जीनोम, उसकी अनुक्रमण, विश्लेषण और जीवों की जीवविज्ञान को समझने में इसके प्रभाव से संबंधित अवधारणाओं का विस्तार से वर्णन किया गया है। कक्षा ग्यारहवीं के अध्याय 7 में छात्र पहले ही यह अध्ययन कर चुके हैं कि आनुवंशिक सूचना डीएनए में कैसे संकेतित होती है और प्रक्रियाएँ, जैसे ट्रांसक्रिप्शन और ट्रांसलेशन।
5.1 जीनोम का मानचित्रण: आनुवंशिक और भौतिक
किसी जीव का जीनोम उसके कोशिका में उपस्थित सम्पूर्ण आनुवंशिक सूचना होती है। प्रोकैरियोट्स के लिए इसमें न्यूक्लॉएड क्षेत्र और प्लाज्मिड में उपस्थित डीएनए शामिल है (जैसा कि जीवाणुओं में होता है); और यूकैरियोट्स के लिए इसमें नाभिक के गुणसूत्र में तथा अंगकोशिकाओं, जैसे माइटोकॉन्ड्रिया और प्लास्टिड्स, उदाहरणार्थ प्रोटिस्ट और सभी बहुकोशिकीय जीवों में उपस्थित डीएनए शामिल होता है (चित्र 5.1)।

चित्र 5.1: जीनोम प्रोकैरियोट्स या यूकैरियोट्स में मौजूद डीएनए सामग्री को दर्शाता है।
कई जीवविज्ञानियों के लिए एक महत्वपूर्ण कार्य यह है कि वे विभिन्न उद्देश्यों के लिए दो जीवों के जीनोम या डीएनए की तुलना करें, जिनमें संरक्षित जीन या डीएनए अनुक्रमों और संबंधों को समझना आदि शामिल है। सरल शब्दों में, किसी जैविक जीव के कई नमूनों से, एक वैज्ञानिक यह जानना चाहता है कि क्या दो या अधिक स्रोतों से आए डीएनए नमूने समान हैं या भिन्न हैं। यदि दो स्रोतों के डीएनए भिन्न हैं, तो ये एक-दूसरे से किस प्रकार भिन्न हैं। आगे, यदि कोई रोचक फ़ीनोटाइप देखा जाता है, तो वैज्ञानिक यह भी जानने में रुचि रख सकता है कि किसी विशेष फ़ीनोटाइप का आनुवंशिक आधार (जीन और उसमें उत्पन्न उत्परिवर्तन) क्या है। इसलिए, ऐसे प्रश्नों के उत्तर देने के लिए एक तकनीकों का समूह आवश्यक है। आदर्श रूप से, यदि हम प्रत्येक नमूने का पूरा डीएनए अनुक्रम जानते हैं, तो हम उनकी तुलना कर सकते हैं और ऐसे सभी प्रश्नों के उत्तर दे सकते हैं।

चित्र 5.2: जीनोम मानचित्र-(a) प्रोकैरियोटिक/ऑर्गेनेल जीनोम का मानचित्र। (b) यूकैरियोटिक जीनोम का मानचित्र, जहाँ $a, b, c, d, e, f, g, h$ जीन लोकस या भौतिक स्थल जैसे SSLP, STS हैं जिन्हें आनुवंशिक दूरी के आधार पर व्यवस्थित किया गया है
जीनोम अनुसंधान में, किसी जीव के जीनोम का मानचित्र तैयार करना अत्यंत महत्वपूर्ण और निर्णायक होता है, जीन या लोकस की पहचान और सापेक्ष स्थिति के दृष्टिकोण से। जीनोम के मानचित्र दो भिन्न दृष्टिकोणों से बनाए जा सकते हैं—आनुवंशिक दृष्टिकोण और भौतिक दृष्टिकोण—जो जैविक जीवों में जीनोम मानचित्रण के लिए क्रमशः आनुवंशिक और भौतिक मानचित्रण तकनीकों को जन्म देते हैं (चित्र 5.2)।
5.1.1 आनुवंशिक मानचित्रण
आनुवंशिक मानचित्रण प्रयोग हमें किसी ज्ञात लक्षण-समूह के लिए उत्तरदायी दो या अधिक आनुवंशिक लोकसों के बीच की दूरी का अनुमान देते हैं। आनुवंशिक मानचित्र, जैसा कि चित्र 5.2 में दिखाया गया है, क्रॉसओवर विश्लेषण करके बनाया जाता है। आपने कक्षा XI में क्रॉसिंग ओवर की घटना पहले ही पढ़ी है (अध्याय 6, खंड 6.2 देखें)। दो या अधिक जीनों या लोकसों के बीच क्रॉसिंग ओवर की दर (पुनःसंयोजन आवृत्ति भी कहा जाता है) का उपयोग उनके बीच की सापेक्ष आनुवंशिक दूरी मापने में सफलतापूर्वक किया गया है। एक मानचित्र इकाई एक प्रतिशत प्रेक्षित क्रॉसओवर (पुनःसंयोजी) के बराबर होती है। सेंटीमोर्गन (cM) इकाई का उपयोग संतति के लक्षण आवृत्ति के आधार पर जीनों के बीच आनुवंशिक दूरी दर्शाने के लिए किया जाता है। दो चिह्नों (लोकसों) के पुनःसंयोजन द्वारा विपृथकीकरण की संभावना उनके बीच की दूरी के अनुक्रमानुपाती होती है। इसलिए, घटती पुनःसंयोजन आवृत्ति इस बात का संकेत है कि कहें गए चिह्नों के बीच की दूरी कम है। जब आनुवंशिक लोकसों को उनके बीच की आनुवंशिक दूरियों के आधार पर व्यवस्थित किया जाता है, तो यह जीव का आनुवंशिक मानचित्र बनाता है। इस दृष्टिकोण का मुख्य दोष यह है कि किसी ज्ञात लक्षण-समूह के लिए उत्तरदायी सीमित संख्या में आनुवंशिक लोकस ही उपलब्ध होते हैं। इससे आनुवंशिक मानचित्र विरल या मोटे दाने वाले बनते हैं और इसलिए ऐसे मानचित्रों की नई लक्षणों को जीनोम में सूक्ष्म मानचित्रण के लिए उपयोगिता सीमित हो जाती है।
5.1.2 भौतिक मानचित्रण
जीनोम के भौतिक मानचित्रण (physical mapping) एक विकल्प है, जिसमें डीएनए आधारित मानचित्र लक्षणों का उपयोग करके जीनोम पर विशिष्ट स्थानों की पहचान की जाती है। प्रतिबंध एंजाइम स्थलों (restriction mapping), सरल अनुक्रम लंबाई बहुरूपता (SSLP), और अनुक्रम टैग स्थलों (STS) की उपस्थिति को मानचित्र लक्षणों के रूप में उपयोग किया गया है (चित्र 5.2)। ये लक्षण जीनोम का उच्च संकल्प भौतिक मानचित्र बनाने में सहायक होते हैं। आइए इन जीनोम मानचित्र लक्षणों को संक्षेप में समझें।
प्रतिबंध अंश लंबाई बहुरूपता (RFLP): RFLP एक मानचित्रण तकनीक है जो एक DNA एंडोन्यूक्लिएस एंजाइम, प्रतिबंध एंजाइम (अनुच्छेद 3.3, अध्याय 3 देखें) का उपयोग करती है जो DNA को एक विशिष्ट आधार अनुक्रम पर काटता है जिससे DNA का खंडन होता है। पचाए गए DNA के खंडों का विखंडन प्रतिरूप तब दिखाई देता है जब खंडों को उनके आकार के आधार पर एगरोज जेल इलेक्ट्रोफोरेसिस द्वारा अलग किया जाता है। आमतौर पर, वृत्ताकार DNA में एक एकल कट एक DNA खंड देता है, जबकि दोहरा कट दो खंडों को जन्म देता है। रेखीय DNA के लिए, एक एकल कट दो DNA खंडों का परिणाम देता है जबकि दोहरा कट तीन खंडों को देता है। DNA को कुछ एंजाइमों जैसे EcoRI, BamHI आदि के साथ या तो एकल रूप से या क्रमिक रूप से पचाया जाता है ताकि प्रतिबंध स्थलों का एक मानचित्र बनाया जा सके जो वे उत्पन्न करने वाले खंडों के आकार पर आधारित होता है। यह अभ्यास जीनोम के प्रतिबंध स्थलों का एक उच्च-रिज़ॉल्यूशन भौतिक मानचित्र देता है। ऐसा मानचित्र आसानी से अन्य मानचित्रों से तुलना किया जा सकता है जिसमें कुछ प्रतिबंध स्थलों के लाभ या हानि को देखकर DNA नमूनों में प्रेक्षित प्रतिबंध अंश लंबाई बहुरूपता को खंडों की लंबाई में आए बदलावों द्वारा नोट किया जाता है (चित्र 5.3)। उदाहरण के लिए, किसी रोग के इतिहास वाले परिवार में, रोगग्रस्त और रोग-रहित सदस्यों के बीच इन मानचित्रों की तुलना से रोग से संबंधित किसी विशिष्ट RFLP लोकस की पहचान में मदद मिल सकती है। सहयोग मानचित्रण जनसंख्याओं के बीच आनुवंशिक विचलन की पहचान में भी सहायक हो सकता है। हालांकि, यह भी महत्वपूर्ण है कि एक स्तनधारी जीनोम में लगभग 10^5 विभिन्न प्रतिबंध एंजाइम स्थल हो सकते हैं और यह काफी संभव है कि EcoRI जैसा कोई प्रतिबंध एंजाइम एक स्तनधारी जीनोम को लाखों खंडों में काट दे और इन्हें इलेक्ट्रोफोरेसिस के आधार पर अलग नहीं किया जा सकता। इस समस्या को सदर्न संकरण तकनीक द्वारा संबोधित किया जाता है जिसमें विशिष्ट DNA प्रोब

आकृति 5.3: भौतिक जीनोम मानचित्रों की तुलना। दोनों मानचित्र स्थल $c$ पर भिन्न हैं। प्रतिबंध स्थल $c$ जीनोम (2) में अनुपस्थित है या खो गया है जीनोम (1) की तुलना में या प्रतिबंध स्थल $c$ जीनोम (1) में उपस्थित है या प्राप्त हुआ है जीनोम (2) की तुलना में। एक प्रोकैरियोटिक/ऑर्गेनेल जीनोम (1) और जीनोम (2) का भौतिक मानचित्र, जहाँ a, b, c, $d$, e, $f$ भौतिक स्थल हैं (जैसे प्रतिबंध एंजाइम स्थल) जो भौतिक दूरियों के आधार पर व्यवस्थित हैं, (3) जीनोम (1) और जीनोम (2) का RFLP पैटर्न जो दोनों जीनोमों के प्रतिबंध पाचन और एगरोज जेल इलेक्ट्रोफोरेसिस पर अलगाव द्वारा प्राप्त किया गया है। जीनोम (1) में प्रतिबंध स्थल $c$ की उपस्थिति (x) में दो छोटे अद्वितीय बैंडों के परिणामस्वरूप देती है जबकि जीनोम (2) में प्रतिबंध स्थल $c$ की अनुपस्थिति (y) में एक बड़ा अद्वितीय बैंड देती है।

आकृति 5.4: प्रतिबंध खंड लंबाई बहुरूपता (RFLP) का उपयोग करते हुए भौतिक मानचित्रण-(a) एक प्रोब P दो DNA मॉर्फ़ का पता लगाता है जब DNA को एक निश्चित प्रतिबंध एंजाइम (RE) द्वारा काटा जाता है। (b) प्रमुख रोग फ़नोटाइप D वंशावली दर्शाती है कि D लोकस RFLP लोकस से लिंक्ड है; केवल बच्चा 8 पुनर्संयोजी है
सरल अनुक्रम लंबाई बहुरूपता (SSLP) मानचित्र: एक बड़ा जीनोम विभिन्न पुनरावृत्ति अनुक्रमों की सरणियाँ रखता है, जिनकी लंबाई भिन्न-भिन्न हो सकती है। सूक्ष्मउपग्रह और लघुउपग्रह ऐसे पुनरावृत्ति अनुक्रम हैं जिनका उपयोग जीनोम के भौतिक मानचित्रण के लिए इसकी लंबाई प्रकार के आधार पर किया जा सकता है (इस पुस्तक के अध्याय 4 की धारा 4.1 देखें)।
अनुक्रम टैगित साइट्स (STS) का उपयोग कर भौतिक मानचित्रण: अनुक्रम टैगित साइट्स भौतिक मानचित्रण में एक उपयोगी संवर्धन हैं। STS अद्वितीय डीएनए अनुक्रम $(200-500 \mathrm{bp})$ होते हैं जो जीनोम में एक बार होते हैं और जिनका जीनोम में अद्वितीय स्थान ज्ञात है। किसी दिए गए डीएनए खंड में STS साइट की उपस्थिति या अनुपस्थिति को बहुलीकरण श्रृंखला अभिक्रिया (PCR) का उपयोग कर आसानी से दिखाया जा सकता है। इसलिए, इन साइटों की उपस्थिति जीनोम के भौतिक मानचित्र के निर्माण में एक उपयोगी स्थान चिन्ह के रूप में कार्य करती है। इन्हें प्रायः प्रतिबंधन मानचित्रों के साथ मिलाया जाता है ताकि संघ मानचित्रण के संकल्प और प्रभावशीलता में सुधार हो सके।
5.2 उच्च-थ्रूपुट डीएनए अनुक्रमण
जबकि हम भौतिक और आनुवंशिक मानचित्र की तुलना करके जीवों को उनके आनुवंशिक या भौतिक मानचित्र के आधार पर भिन्न-भिन्न और वर्गीकृत कर सकते हैं, किसी जीव का पूरा डीएनए अनुक्रम प्राप्त करना अत्यधिक वांछनीय है। पहले के दिनों में, पूरे जीनोम का अनुक्रमण अत्यधिक महँगा था और इसलिए कम खर्चीली तकनीकों का उपयोग यह सुनिश्चित करने के लिए किया जाता था कि रुचि का क्षेत्र उस डीएनए के छोटे लोकस तक सीमित हो जो रुचि के लक्षण से सांख्यिकीय रूप से संबद्ध है। पूरे जीनोम अनुक्रमण की अत्यधिक लागत से बचने के लिए केवल इस छोटे रुचि के क्षेत्र का ही अनुक्रमण किया जाता था। हालाँकि, इस दृष्टिकोण के लिए एक ऐसे जीनोम का मानचित्र आवश्यक होता है जिसे संदर्भित या अन्य लोगों से तुलना किया जा सके। अनुक्रमण तकनीक में प्रगति ने अब यह संभव बना दिया है कि किसी भी जीव का पूरा डीएनए अनुक्रम प्राप्त किया जा सके, यदि हम उसका डीएनए प्राप्त कर सकें। प्रारंभ में, प्रति आधार डीएनए अनुक्रमण की लागत मानव सहित यूकेरियोटिक जीवों के पूरे डीएनए अनुक्रमण को करने के लिए अत्यधिक थी। हालाँकि, वर्तमान में यह काफी बदल गया है। इस खंड में, हम देखेंगे कि पिछले तीन दशकों में डीएनए अनुक्रम तकनीक कैसे विकसित हुई है।
5.2.1 प्रथम पीढ़ी की डीएनए अनुक्रमण तकनीक
जीनोम अनुक्रमण का प्रारंभिक या प्रथम चरण एक विस्तृत और बहु-चरणीय प्रक्रिया थी। इसमें पल्स फील्ड जेल इलेक्ट्रोफोरेसिस (PFGE) का उपयोग करके अगरोस जेल पर गुणसूत्र को पृथक करना शामिल था। पृथक किए गए गुणसूत्र को तब प्रतिबंधन पाचन (restriction digestion) के अधीन किया गया और पचे हुए खंडों को उच्च-क्षमता वाले क्लोनिंग वेक्टरों, जैसे BAC, YAC, PAC आदि से जोड़ा गया (अध्याय 2, खंड 2.3 देखें)। इन क्लोनों में उपस्थित DNA अक्सर एक-दूसरे के ओवरलैपिंग होते हैं और ऐसे प्रयासों से प्राप्त क्लोनों को न्यूनतम क्लोनों के एक समूह में व्यवस्थित किया जा सकता है जो जीव से ओवरलैपिंग DNA के समुच्चय को वहन करते हैं।
इन क्लोनों से DNA खंडों को भौतिक रूप से मैप किया जाता है और उन्हें सीक्वेंसिंग प्लाज्मिड में सब-क्लोन किया जाता है ताकि प्रथम पीढ़ी की DNA अनुक्रमण तकनीकों का उपयोग करके क्लोन किए गए DNA का अनुक्रमण किया जा सके।
पहली पीढ़ी की डीएनए अनुक्रमण तकनीक में श्रृंखला समापन द्वारा अनुक्रमण (कक्षा XI जैवप्रौद्योगिकी पाठ्यपुस्तक की धारा 12.9.1 देखें) नामक विधि का उपयोग किया गया। इस विधि में, एकल डोरी वाले डीएनए की एक सीढ़ी बनाई जाती है, जिसमें प्रत्येक एक विशिष्ट आधार (जिसे समापन आधार से जुड़े विशिष्ट फ्लोरेसेंस से पहचाना जाता है) स्थान पर समाप्त होता है। इन खंडों को उनके आकार के आधार पर कैपिलरी जेल इलेक्ट्रोफोरेसिस तकनीक का उपयोग करके अलग किया जाता है (जिसमें आवेशित अणु अर्थात् डीएनए खंडों की इलेक्ट्रोफोरेसिस जेल से भरी कैपिलरी में की जाती है)। कैपिलरी के निचले सिरे के पास एक फ्लोरेसेंस डिटेक्टर रखा जाता है जो डिटेक्टर के सामने से गुजरने वाले एकल डोरी वाले डीएनए का फ्लोरेसेंस पढ़ता है। इस तकनीक का आउटपुट फ्लोरेसेंस का एक क्रोमैटोग्राम होता है, जहाँ प्रत्येक चोटी एक आधार स्थान को दर्शाती है और चोटी का रंग बताता है कि वहाँ कौन-सा डीएनए आधार मौजूद है। एकल चलन में प्रत्येक कैपिलरी लगभग 800-1000 आधार लंबे डीएनए का अनुक्रमण करती है। एक एकल डीएनए अनुक्रमक में 96 कैपिलरीज़ (जिनमें 96 भिन्न अनुक्रमण प्रतिक्रियाएँ लोड की जाती हैं) हो सकती हैं जो एक मशीन में समानांतर रूप से कार्य करती हैं। इस प्रकार की व्यवस्था प्रति चलन, प्रति मशीन 96000 आधार तक प्रदान कर सकती है।
इस तकनीक का एक दोष यह है कि यह समय लेने वाली और श्रम-साध्य है जिससे जीनोम अनुक्रमण की लागत निषेधात्मक रूप से अधिक हो जाती है। इस चुनौती को दूर करने के लिए, डीएनए अनुक्रमण तकनीक की अगली पीढ़ी विकसित की गई।
5.2.2 अगली पीढ़ी (दूसरी) डीएनए अनुक्रमण तकनीक
अगली पीढ़ी की डीएनए अनुक्रमण तकनीक एक बड़े पैमाने पर समानांतर (एक मशीन में लाखों समानांतर अनुक्रमण प्रतिक्रियाएँ) डीएनए अनुक्रमण दृष्टिकोण का उपयोग करती है। इस तकनीक में समय लेने वाली क्लोनिंग और सब-क्लोनिंग चरणों की आवश्यकता नहीं होती है और इसलिए, जीनोम अनुक्रमण परियोजना के लिए आवश्यक लागत को कम करती है। पहली पीढ़ी की अनुक्रमण विधियों की तुलना में, अगली पीढ़ी की विधियाँ सरल कार्यप्रवाह, उच्च डेटा सटीकता (उच्च कवरेज की गहराई के कारण) और व्यापक अनुप्रयोग सीमा जैसे लाभ प्रदान करती हैं।
हालाँकि इस रणनीति के कई दृष्टिकोण हैं, हम सबसे लोकप्रिय अगली पीढ़ी की अनुक्रमण तकनीक प्लेटफॉर्म में से एक, अर्थात् इलुमिना अनुक्रमण तकनीक (चित्र 5.5) के कार्य को देखेंगे। यह एक फ्लो सेल (कांच की स्लाइड) का उपयोग करती है जहाँ डीएनए खंडों को ब्रिज पीसीआर प्रवर्धन नामक विशेष विधि के माध्यम से प्रवर्धित किया जाता है। एक फ्लो सेल एक ठोस आधार है, जहाँ एकल स्ट्रैंड छोटे ओलिगोन्यूक्लियोटाइड्स ($8-10$ आधार) जुड़े होते हैं। प्रारंभ में, डीएनए को छोटे टुकड़ों (1-2 केबी) में खंडित किया जाता है और इन डीएनए खंडों से छोटे ओलिगोन्यूक्लियोटाइड अडैप्टर जुड़े होते हैं। ये अडैप्टर फ्लो सेल पर ओलिगोन्यूक्लियोटाइड्स के पूरक होते हैं और इसलिए, डीएनए खंडों को फ्लो सेल से जोड़ने में मदद करते हैं। इन अडैप्टरों का उपयोग प्राइमर के रूप में किया जाता है और नई डीएनए स्ट्रैंड्स संश्लेषित की जाती हैं। इस प्रक्रिया के दौरान, खंड पुल जैसी संरचना बनाते हैं क्योंकि अडैप्टर फ्लो सेल से जुड़े ओलिगोन्यूक्लियोटाइड्स के पूरक होते हैं। इसलिए, इसे ब्रिज प्रवर्धन कहा जाता है।
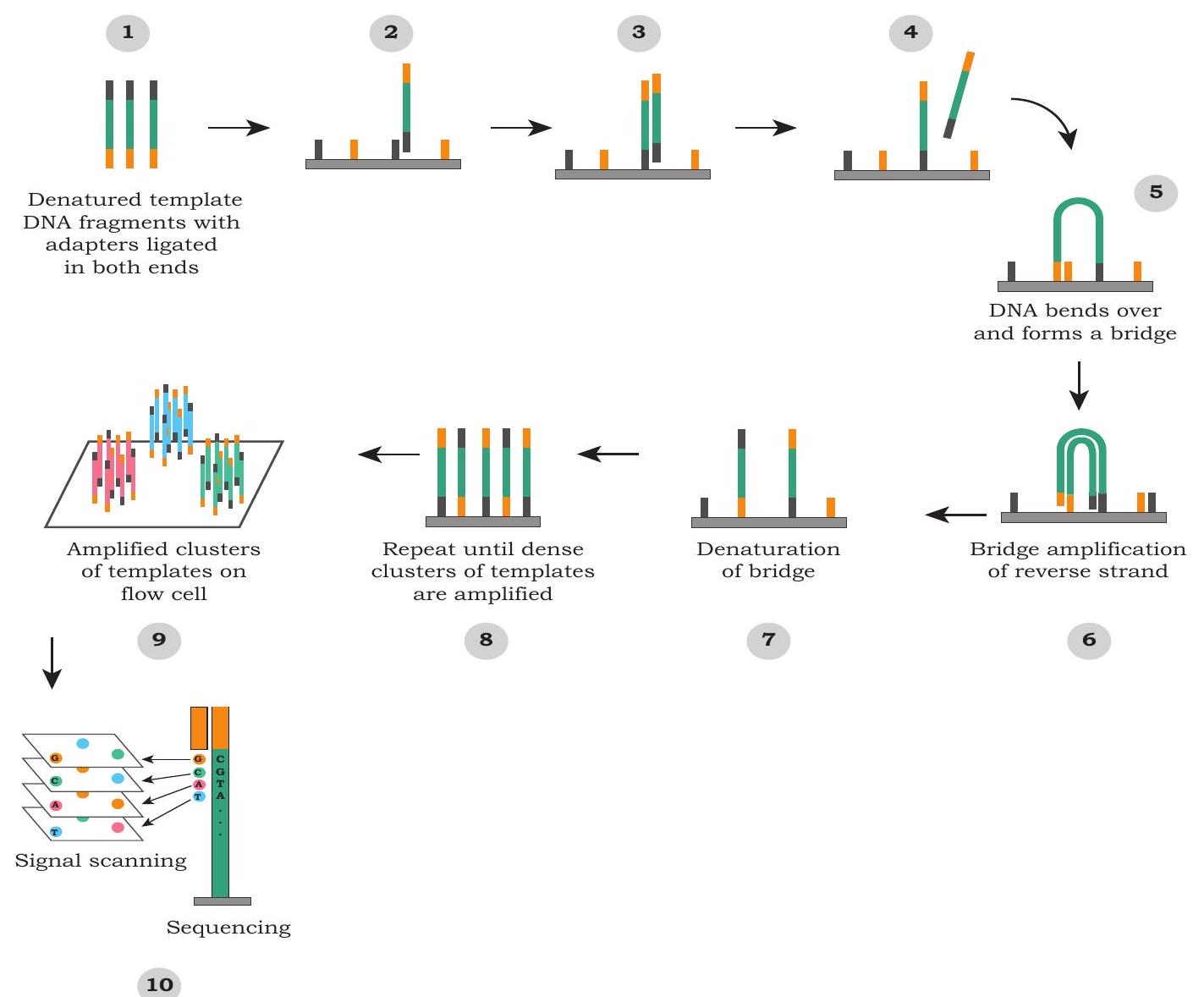
चित्र 5.5: एक लोकप्रिय नेक्स्ट जनरेशन सीक्वेंसिंग तकनीक
इससे एक ही स्थान पर समान फ्रैगमेंट्स का एक समूह बनता है। पीसीआर एम्प्लिफिकेशन के बाद, सीक्वेंसिंग प्राइमर का उपयोग किया जाता है, जो एक एडाप्टर से बाइंड होता है और प्रत्येक फ्रैगमेंट को एक ही समय पर सीक्वेंस किया जाता है। सीक्वेंसिंग के लिए, फ्लोरोसेंटली टैग किए गए dNTPs का उपयोग कॉम्प्लिमेंट्री स्ट्रैंड के संश्लेषण के लिए किया जाता है और प्रत्येक बेस यानी dATPs, dTTPs, dGTPs या dCTPs, को अलग-अलग फ्लोरोसेंट टैग से जोड़ा जाता है। एक समय पर केवल एक ही बेस जोड़ा जाता है और शामिल किए गए बेस को उसके फ्लोरोसेंस के प्रकार के आधार पर पढ़ा जाता है। इसके बाद अगला बेस जोड़ा जाता है और फ्लोरोसेंस इमेज्ड की जाती है। इस तरह, लाखों फ्रैगमेंट्स एक ही समय में समानांतर रूप से सीक्वेंस किए जाते हैं।
अनुक्रमण प्रतिक्रिया पॉलिमरेज़ आधारित है और यह प्रवाह कोशिका में लेबलयुक्त आधार (इन आधारों में अद्वितीय प्रतिदीप्ति टैग होता है) को पास करके की जाती है। ये प्रतिदीप्त आधार उस डीएनए टेम्पलेट के आधार पर शामिल होंगे जो प्रारंभ में प्रवाह कोशिकाओं से जुड़ा होता है। नव-शामिल आधार की प्रतिदीप्ति प्रवाह कोशिकाओं पर जुड़े डीएनए खंड का अनुक्रम प्रकट करती है (चित्र 5.5)। प्रवाह कोशिका की छवि रिकॉर्ड करने के बाद, शामिल आधार का प्रतिदीप्त टैग हटा दिया जाता है और फिर अगला आधार प्रवाह कोशिका के माध्यम से पास किया जाता है (चित्र 5.5)। संपूर्ण अनुक्रमण को प्रतिदीप्त छवियों की सरणी के रूप में रिकॉर्ड किया जाता है, जहाँ डीएनए अनुक्रमन को किसी दिए गए निर्देशांक या स्थान पर प्रतिदीप्ति परिवर्तनों के रूप में रिकॉर्ड किया जाता है। ऐसी छवियों की सरणी लाखों ऐसे स्थानों का अनुक्रम समानांतर में प्रकट करती है।
यद्यपि यह तकनीक लाखों डीएनए को समानांतर में अनुक्रमित करने में सक्षम है, यह पहली पीढ़ी की तकनीक की तुलना में बहुत छोटे (75 से 300 आधार लंबे) अनुक्रम उत्पन्न करती है। हालांकि, अतिरिक्त अनुक्रमण रणनीतियाँ उपलब्ध हैं जो लघु अनुक्रमण आउटपुट द्वारा प्रस्तुत कुछ सीमाओं को दूर करने के लिए हैं।
5.2.3 डीएनए अनुक्रमण तकनीक में कुछ हालिया प्रगति
डीएनए अनुक्रमण तकनीक में एक हालिया प्रगति नैनोपोर का उपयोग है। इसे तीसरी पीढ़ी की अनुक्रमण तकनीक भी माना जाता है। यहाँ, अनुक्रमण तब किया जाता है जब एकल डोरी वाले डीएनए का कोई विशिष्ट आधार नैनोपोर से गुजरता है तो विद्युत धारा में आने वाले बदलाव को मापा जाता है (चित्र 5.6)। इस तकनीक के वर्तमान कार्यान्वयन में दो प्रोटीनों का उपयोग होता है—एक डीएनए हेलिकेज और एक पोरिन-जैसा अणु जो एक संश्लेषित झिल्ली पर नैनोपोर बनाने के लिए इकट्ठा होता है और अनुक्रमण सेल को दो डिब्बों में बाँटता है। डीएनए हेलिकेज द्वि-डोरी वाले डीएनए को पकड़ता है और उसे खोलता है तथा उसकी एक डोरी को नैनोपोर से धकेलता है। नैनोपोर के पार एक आयनिक धारा बनाए रखी जाती है और उसे लगातार निगरानी में रखा जाता है। जब डीएनए के आधार छिद्र से गुजरते हैं, तो पोरिन-जैसा अणु एकल डोरी वाले डीएनए के आधारों के साथ भिन्न-भिन्न रूप से संपर्क करता है और आयनिक धारा को बाधित करता है। प्रत्येक आधार द्वारा उत्पन्न बाधा बहुत विशिष्ट होती है और विद्युत धारा में आने वाले इन संकेतों को मापकर संगत आधारों की पहचान की जा सकती है। नैनोपोर आधारित अनुक्रमण तकनीज़ तेज़ और सरल नमूना प्रसंस्करण प्रदान करती है और इसमें वास्तविक समय में अनुक्रमण परिणाम दिखाने की क्षमता है, जिससे यह मैदानी सेटअप में भी जीनोटाइपिंग के लिए अत्यधिक उपयोगी हो जाती है। नैनोपोर अनुक्रमण का एक अन्य प्रमुख लाभ अत्यधिक लंबे अनुक्रमण रीड्स (जहाँ तक $1 \mathrm{Mb}$ या इससे भी अधिक) का अपेक्षाकृत कम लागत पर उत्पादन है।
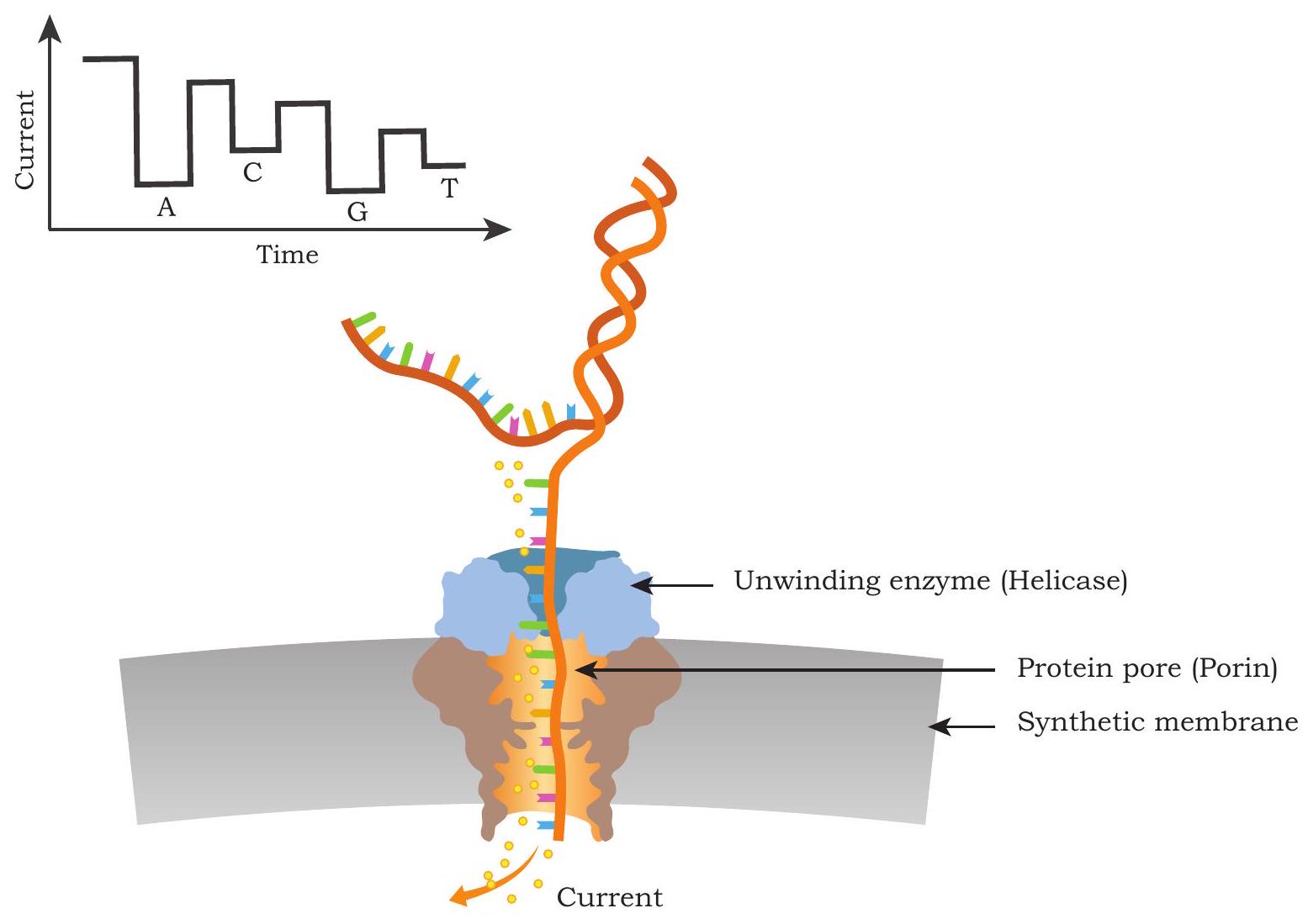
चित्र 5.6: नैनोपोर आधारित अनुक्रमण तकनीक। पीले बिंदु एक छिद्र के माध्यम से बहने वाले आयन हैं जो प्रोटीन छिद्र (नीले रंग) द्वारा बनाए गए नैनोपोर में धारा उत्पन्न करते हैं। प्रत्येक बेस के नैनोपोर से गुजरने से आयनिक धारा में विशिष्ट परिवर्तन होते हैं। इन आयनिक धारा में परिवर्तनों को मापा जाता है और संगत बेस की पहचान की जाती है। यहां पढ़ा गया अनुक्रम ACGT है। अनवाइंडिंग एंजाइम कुशलता से डीएनए को पकड़ता है, प्रोटीन छिद्र पर डॉक करता है और डीएनए की एक स्ट्रैंड को नैनोपोर के माध्यम से चलाता है।
5.3 अन्य जीनोम-संबंधी प्रौद्योगिकियाँ
जीनोम अनुक्रमण कई स्वादों या विविधताओं में आता है। इसमें संपूर्ण जीनोम अनुक्रमण (WGS), लक्षित अनुक्रमण, मेटाजीनोमिक्स आदि शामिल हैं, जैसा कि निम्नलिखित खंडों में वर्णित है।
5.3.1 संपूर्ण जीनोम अनुक्रमण (WGS)
WGS एक व्यापक विधि है जिससे किसी जीव के सम्पूर्ण जीनोम की डीएनए अनुक्रम निर्धारित की जाती है। WGS का उपयोग करके हीमोफिलस इन्फ्लुएंज़े का पहला जीनोम अनुक्रमित किया गया था। सम्पूर्ण जीनोम अनुक्रमों की उपलब्धता वंशानुगतिकी के आनुवंशिक नियमन को समझने, वंशानुगत आनुवंशिक विकारों की पहचान करने आदि में सहायक होती है। मानव जीनोम का सम्पूर्ण जीनोम अनुक्रमण 1990 में प्रारम्भ हुआ और पूर्ण अनुक्रम 2003 में प्रकाशित हुआ। अनुक्रमण प्रौद्योगिकियों में प्रगति (जैसा कि पिछले खण्ड में चर्चा की गई है) के साथ अब सम्पूर्ण जीनोम को कुछ ही दिनों में अनुक्रमित करना सम्भव है। बड़ी संख्या में सूक्ष्मजीवों, जानवरों और पौधों का सम्पूर्ण जीनोम अनुक्रम पहले ही प्राप्त किया जा चुका है। WGS की उपलब्धता मानव रोगों के उपचार के लिए वैयक्तिकृत योजनाएँ बनाने में सहायक हो सकती है या कैंसर कोशिकाओं में विचरणों को पढ़कर चिकित्सक को सर्वोत्तम कीमोथेरेपी चुनने में मदद कर सकती है।
जब सम्पूर्ण जीनोम अनुक्रमण एक संदर्भ जीनोम अनुक्रम (पहले से अनुक्रमित और आनुवंशिक रूप से सम्बन्धित जीनोम अनुक्रम) का उपयोग करके अपने व्यक्तिगत अनुक्रमण रीड्स को जीनोम अनुक्रम में जोड़ता है, तो इसे संदर्भ-आधारित जीनोम अनुक्रमण कहा जाता है। जब ऐसा कोई संदर्भ उपयोग नहीं किया जाता है, तो इसे डी नोवो जीनोम अनुक्रमण कहा जाता है।
5.3.2 लक्षित अनुक्रमण
लक्षित अनुक्रमण परियोजना इस जानने के लिए की जाती है कि जीनोम के लक्षित क्षेत्रों में विचरण है या नहीं। ऐसी परियोजनाएँ पहले से अनुक्रमित जीनोम को संदर्भ जीनोम अनुक्रम के रूप में उपयोग करती हैं ताकि नव अनुक्रमित जीनोम को असेंबल किया जा सके। लक्षित अनुक्रमण जीनोम के चयनित जीनों या जीनोमिक क्षेत्रों के अनुक्रमण और विश्लेषण पर केंद्रित होता है। इसका उपयोग विभिन्न जीनोमों के बीच जीनोमिक क्षेत्रों के छोटे समूह की तुलना करके उत्परिवर्तन, सम्मिलन या विलोपन जैसे विचरणों की पहचान के लिए किया जा सकता है। इस उद्देश्य के लिए, लक्षित जीनोमिक क्षेत्रों का चयन किया जाता है, उन्हें पृथक किया जाता है और फिर अनुक्रमित किया जाता है। आमतौर पर इस उद्देश्य के लिए PCR का उपयोग किया जाता है।
क्लिनिकल एक्सोम अनुक्रमण भी लक्षित पुनःअनुक्रमण के समान ही होता है। क्लिनिकल एक्सोम अनुक्रमन उन जीनों को कवर करता है जो रोग-संबंधी होने के लिए जाने जाते हैं (जैसे कि OMIM, HGMD आदि डेटाबेसों से रिपोर्ट और क्यूरेट किए गए हैं)। क्लिनिकल एक्सोम अनुक्रमण पर आधारित आनुवंशिक निदान परीक्षण, पूर्ण जीनोम अनुक्रमण की तुलना में लागत प्रभावी होते हैं।
उच्च-थ्रूपुट DNA अनुक्रमण के विकास के साथ, DNA अनुक्रमण प्रौद्योगिकी स्वयं को किसी जीव का केवल जीनोम अनुक्रम प्रकट करने से कहीं अधिक करने के लिए अनुकूलित किया गया है। कोई इस प्रौद्योगिकी का उपयोग किसी जीव के जीव विज्ञान को जीनोम स्तर पर अध्ययन करने के लिए कर सकता है। अनुक्रमण प्रौद्योगिकियों की कुछ उन्नत अनुप्रयोग ChipSeq, RNASeq आदि हैं।
ChipSeq सिक्वेंसिंग प्रोजेक्ट्स का उपयोग किसी ट्रांसक्रिप्शन रेगुलेटर के जीनोम-व्यापी DNA बाइंडिंग साइट्स को मैप करने के लिए किया जाता है। चूँकि ट्रांसक्रिप्शन रेगुलेटर एक या अधिक जीनों की जीन अभिव्यक्ति को नियंत्रित करने में महत्वपूर्ण भूमिका निभाते हैं, ChipSeq प्रोजेक्ट्स किसी ट्रांसक्रिप्शन रेगुलेटर के जीनोम-व्यापी DNA बाइंडिंग साइट्स (साथ ही लक्ष्य जीनों) को जानने के लिए उपयोग किए जाते हैं।
RNASeq प्रोजेक्ट्स किसी जीव, ऊतक या नमूने की वैश्विक जीन अभिव्यक्ति प्रोफ़ाइल (ट्रांसक्रिप्टोम) का अध्ययन करने के लिए किए जाते हैं। लक्ष्य नमूने से कुल RNA निकाला जाता है और cDNA संश्लेषित किया जाता है, जिसे नेक्स्ट जनरेशन सिक्वेंसिंग तकनीकों का उपयोग करके सिक्वेंस किया जाता है।
इस प्रकार उत्पन्न सिक्वेंसिंग रीड्स जीनोम पर विभिन्न जीनों से मैप किए जाते हैं और उन जीनों से मैप होने वाले ऐसे रीड्स की गिनती उनकी अभिव्यक्ति के अनुपात में होती है।
5.3.3 मेटाजीनोमिक्स: किसी सूक्ष्मजीव समुदाय में मौजूद DNA या cDNA का सिक्वेंसिंग
मेटाजीनोमिक्स परियोजनाओं का उपयोग जीवों के समुदाय के जीनोमिक्स का अध्ययन करने के लिए किया जाता है। यहां, हम जीवों को अलग नहीं करते हैं या उन्हें व्यक्तिगत रूप से कल्चर नहीं करते हैं। वास्तव में, मेटाजीनोमिक्स किसी विशेष वातावरण में रहने वाले सूक्ष्मजीवों से सीधे प्राप्त कुल आनुवांशिक पदार्थ का अध्ययन है, जैसे कि पशु आंत में रहने वाले सूक्ष्मजीव या किसी विशेष पारिस्थितिक वातावरण में रहने वाले सूक्ष्मजीव। इसका चिकित्सा सूक्ष्मजीव विज्ञान, कृषि के साथ-साथ पर्यावरणीय सूक्ष्मजीव विज्ञान में कई अनुप्रयोग हैं। विशेष रूप से, यह सूक्ष्मजीव समुदायों की विविधता का अध्ययन करने और यह समझने में मदद करता है कि वातावरण में बदलाव के साथ इन सूक्ष्मजीवों की विविधता कैसे बदलती है। मेटाजीनोमिक्स महत्वपूर्ण औद्योगिक अनुप्रयोगों वाले नए जीनों या एंजाइमों की पहचान करने में भी मदद कर सकता है। उदाहरण के लिए, चरम वातावरणों में रहने वाले सूक्ष्मजीव समुदायों का अध्ययन, जैसे कि उच्च तापमान वाले सल्फर स्प्रिंग्स, उच्च तापमान पर प्रतिरोधी और कार्यात्मक नए एंजाइमों की पहचान करने में मदद कर सकता है।
इस प्रकार, मेटाजीनोमिक्स का उपयोग आंत के सूक्ष्मजीवों, गले के सूक्ष्मजीवों, शौचालय की सीट पर मौजूद सूक्ष्मजीवों और कई अन्य सूक्ष्मजीवों के जीनोम को समझने में किया जाता है। यह विविध रूपों के सूक्ष्मजीवों के जीनोम और उनके सहसंबंधों के बारे में अंतर्दृष्टि प्रदान करता है। उदाहरण के लिए, विभिन्न रूपों के वायरसों के जीनोम के अध्ययन से वायरस-मेज़बान अंतःक्रिया, महामारी विज्ञान और वायरसों के विकास को समझने में मदद मिल सकती है। मेटाजीनोमिक्स नमूने में कई जीनोम की उपस्थिति डेटा विश्लेषण में विशेष चुनौतियाँ पैदा करती है, इसलिए उन्हें कंप्यूटिंग एल्गोरिदम के एक अलग सेट की आवश्यकता होती है।
5.4 जीनोम इंजीनियरिंग
इस तकनीक का उपयोग जीनोम को संशोधित करने के लिए किया जाता है। इसे अक्सर एक या अधिक जीनों को पेश करने या हटाने के लिए किया जाता है। इस इंजीनियरिंग का लक्ष्य एक नई कार्यक्षमता को पेश करना या किसी जैविक जीव की मौजूदा कार्यक्षमता को संशोधित करना (या हटाना) होता है। इस खंड में, हम उन दृष्टिकोणों की एक झलक पाएंगे जिनका उपयोग जीनोम को इंजीनियर करने के लिए किया गया है। दृष्टिकोणों में से एक ट्रांसपोसॉन या कूदने वाले जीनों का उपयोग करता है ताकि किसी जीनोम में किसी जीन(जीनों) को निष्क्रिय या हटाया जा सके। एक अन्य दृष्टिकोण जीनोम को संपादित करना है बिना जीनोम में किसी अनचाहे अतिरिक्त परिवर्तन के।
5.4.1 ट्रांसपोसॉन सम्मिलन द्वारा जीन का नॉक-आउट और नॉक-इन
ट्रांसपोज़ॉन डीएनए अनुक्रम होते हैं जिन्हें ‘जंपिंग जीन्स’ भी कहा जाता है, जो जीनोम पर एक स्थान से दूसरे स्थान पर चले जाते हैं और लगभग सभी जीवों (प्रोकैरियोट्स और यूकैरियोट्स दोनों) में पाए जाते हैं। डीएनए ट्रांसपोज़ॉन आमतौर पर ‘कट और पेस्ट’ तंत्र द्वारा चलते हैं जिसमें ट्रांसपोज़ॉन को एक स्थान से काटकर जीनोम में कहीं और सम्मिलित किया जाता है। कई बार, डीएनए खंड के इस प्रकार के संचलन से सम्मिलन स्थल पर उपस्थित किसी जीन का सम्मिलन निष्क्रियण हो जाता है। ट्रांसपोज़ॉन के इस व्यवहार का उपयोग मौजूदा जीन को बाहर करने के लिए किया जाता है। ऐसी रणनीति में, ट्रांसपोज़ॉन को इंजीनियर किया जाता है ताकि वह लक्ष्य जीन के एक विशिष्ट अनुक्रम को पहचान सके, जहाँ वह सम्मिलित होता है। एक बार ट्रांसपोज़ॉन लक्ष्य स्थल पर सम्मिलित हो जाने के बाद, लक्ष्य जीन की कोडिंग फ्रेम बाधित हो जाती है और इसलिए वह मूल ट्रांस्क्रिप्ट उत्पन्न नहीं कर सकता जो जीन उत्पाद को कोड करने के लिए आवश्यक होता है (चित्र 5.7)।
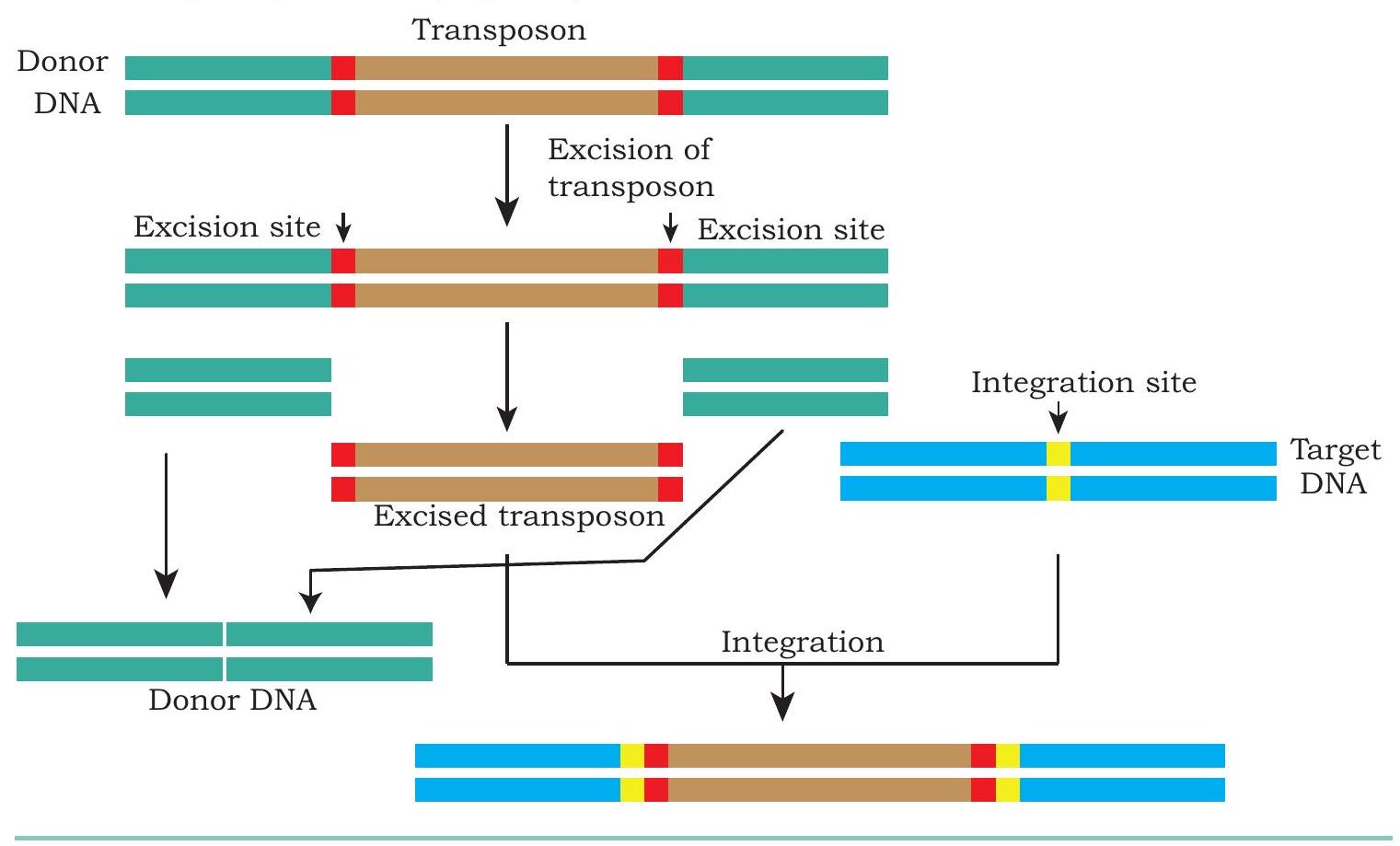
चित्र 5.7: ट्रांसपोज़िशन द्वारा जीनोम इंजीनियरिंग
कुछ विशेष स्थितियों में, जब एक ट्रांसपोज़न किसी विशेष स्थान पर सम्मिलित होता है, तो यह रुचि के जेनेटिक लोकस को बदल देता है, जिससे DNA अनुक्रम जुड़ जाता है जो उस जेनेटिक लोकस पर पहले मौजूद नहीं था। ट्रांसपोज़न का यह गुण किसी कार्यात्मक जीन के नॉक-इन के लिए उपयोग किया जाता है। यह नॉक-इन और नॉक-आउट तकनीक पशु/पादप मॉडलों के निर्माण में मदद करती है ताकि रोग के आण्विक आधार को समझा जा सके और नई दवाओं का विकास किया जा सके।
5.4.2 क्लस्टर्ड रेग्युलरली इंटरस्पेस्ड शॉर्ट पैलिंड्रोमिक रिपीट्स (CRISPR-Cas9) का उपयोग करके जीनोम संपादन
जीनोम संपादन तकनीकों या प्रौद्योगिकियों का एक समूह है जिसका उपयोग किसी जीव के DNA को बदलने के लिए किया जा सकता है—जीनोम के विशिष्ट DNA अनुक्रम को जोड़कर, हटाकर या बदलकर। प्रतिबंधन एंजाइमों की तरह, यह तकनीक जीवाणुओं में प्राकृत रूप से मौजूद जीनोम संपादन प्रणाली का एक जैवप्रौद्योगिकीय रूपांतरण या पुनः उपयोग है। अपने प्राकृतिक संदर्भ में, यह प्रणाली पिछले वायरल हमलों के DNA हस्ताक्षर को याद करने के लिए उपयोग की जाती है ताकि किसी भी बाद के वायरल संक्रमण, जिसमें वायरल DNA पर मिलता-जुलता DNA हस्ताक्षर हो, को प्रभावी रूप से रोका या नष्ट किया जा सके।

आकृति 5.8: CRISPR-Cas9 प्रौद्योगिकी का उपयोग करके जीन संपादन। गाइड RNA Cas9 प्रोटीन कॉम्प्लेक्स के साथ जीनोमिक DNA के विशिष्ट क्षेत्र को स्थिति देता है ताकि डबल स्ट्रैंड ब्रेक प्रेरित किया जा सके। डबल स्ट्रैंड ब्रेक के बाद, DNA अनुक्रम की विलोपन या सम्मिलन द्वारा जीन नॉक-आउट बनाया जा सकता है। जीन संपादन के लिए, एक वांछित म्यूटेंट प्रतिलिपि (ब्रेक साइट के आसपास सुधार/प्रतिस्थापन के लिए) इस क्षेत्र में पेश की जा सकती है ताकि वांछित म्यूटेंट टेम्पलेट DNA की उपस्थिति में समजात पुनर्संयोजन हो सके, जिससे ये उत्परिवर्तन जीनोमिक DNA में एक विशिष्ट स्थान पर प्रस्तुत हो जाएँ।
इस दृष्टिकोण में, एक गाइड RNA को डिज़ाइन किया जाता है और Cas9 एंडोन्यूक्लिएस के साथ कोशिका में उत्पादित किया जाता है। गाइड RNA Cas9 एंडोन्यूक्लिएस को उस स्थान पर स्थिति देने के लिए आवश्यक होता है, जहाँ DNA को संपादित या इंजीनियर किया जाना आवश्यक है। गाइड RNA का एक भाग DNA अनुक्रम के पूरक होता है, जहाँ अनुक्रम को संपादित करने की आवश्यकता होती है। गाइड RNA का दूसरा भाग Cas9 एंडोन्यूक्लिएस को आकर्षित करता है और उससे बंधता है। एक बार यह संपूर्ण कॉम्प्लेक्स (गाइड RNA लक्ष्य DNA और Cas9 से बंधा हुआ) इकट्ठा हो जाता है, तो एंडोन्यूक्लिएस द्वारा DNA में एक डबल स्ट्रैंड ब्रेक प्रस्तुत किया जाता है (आकृति 5.8)। टूटा हुआ DNA एक टेम्पलेट आधारित समजात DNA मरम्मत से गुजरता है, जहाँ एक संशोधित या संपादित DNA टेम्पलेट प्रदान किया जाता है जिसमें वांछित परिवर्तन (संपादित अनुक्रम) होता है। इस प्रकार के डबल स्ट्रैंड ब्रेक और टेम्पलेट-गाइडेड DNA मरम्मत के परिणामस्वरूप, मरम्मत किया गया जीनोमिक DNA अब एक संपादित DNA अनुक्रम रखता है।
बॉक्स 1
CRISPR-Cas9 जेनेटिक कैंची की खोज के लिए 2020 का रसायन शास्त्र में नोबेल पुरस्कार
एमैनुएल शार्पेंटियर, जर्मनी के बर्लिन में मैक्स प्लांक इकाई फॉर द साइंस ऑफ पैथोजन्स में एक शोधकर्ता; और जेनिफर ए. डाउडना, कैलिफोर्निया विश्वविद्यालय, बर्कले में एक जैवरसायनिक, को CRISPR-Cas9 जेनेटिक कैंची, एक जीन-संपादन विधि की खोज के लिए 2020 का रसायन शास्त्र में नोबेल पुरस्कार दिया गया।
CRISPR-Cas9 की अवधारणा प्राकृतिक रूप से होने वाले प्राचीन जीवाणु प्रतिरक्षा तंत्र से संशोधित की गई थी। जीवाणु आक्रमणकारी वायरस के डीएनए को पहचानने और उसे CRISPR अरे कहलाने वाले निष्क्रिय खंडों में काटने के लिए CRISPR-Cas प्रणालियों का उपयोग करते हैं। यह प्रभावी रूप से वायरस को निष्क्रिय कर देता है और जीवाणु की प्रतिरक्षा ‘स्मृति’ में योगदान देता है। 2011 में स्ट्रेप्टोकोकस पायोजेन्स का अध्ययन करते समय, डॉ. शार्पेंटियर ने CRISPR-Cas प्रणाली में tracrRNA नामक एक नई अणु की खोज की और डॉ. डाउडना के साथ CRISPR प्रणाली को पुनःप्रोग्राम करने के लिए सहयोग शुरू किया। साथ में, उन्होंने पाया कि Cas9 प्रोटीन का उपयोग करके CRISPR को लक्ष्य अनुक्रमों पर डीएनए काटने के लिए निर्देशित किया जा सकता है। इससे जीनेटिक सामग्री को बाद में कोशिका की अपनी डीएनए मरम्मत मशीनरी का उपयोग करते हुए इच्छानुसार जोड़ा, हटाया या बदला जा सकता है। शोधकर्ता इस तकनीक का उपयोग जीवित जीव के डीएनए को अत्यधिक उच्च परिशुद्धता के साथ संशोधित करने के लिए कर सकते हैं। यह विशिष्ट जीन संपादन उपकरण जीनेटिक कोड में बहुत छोटे और सटीक परिवर्तन करने की अनुमति देता है, यह माउस मॉडल में समय से पहले बुढ़ापे का कारण बनने वाली बीमारी को रोक सकता है और इलाज किए गए जानवरों का जीवनकाल दोगुना कर सकता है।
5.5 संरचनात्मक, कार्यात्मक और तुलनात्मक जीनोमिक्स
अगली पीढ़ी की अनुक्रमण परियोजनाएँ बड़ी मात्रा में डेटा उत्पन्न करती हैं जिसके विश्लेषण के लिए व्यापक कम्प्यूटिंग की आवश्यकता होती है। कम्प्यूटेशनल जीनोमिक्स एक ऐसा क्षेत्र है जो उच्च प्रदर्शन कम्प्यूटिंग क्लस्टरों और वर्कस्टेशनों का उपयोग करता है ताकि जीनोमिक्स डेटा का विश्लेषण किया जा सके, जिसमें अनुक्रमण प्रौद्योगिकी द्वारा उत्पन्न डेटा भी शामिल है। कम्प्यूटेशनल जीनोमिक्स डीएनए और आरएनए अनुक्रमों के साथ-साथ अन्य प्रायोगिक डेटा पर आधारित जीनोम अनुक्रमों और संबंधित डेटा से जीव विज्ञान को समझने के लिए कम्प्यूटेशनल और सांख्यिकीय विश्लेषण के आधार पर जीनोम के कार्यों को समझने में मदद करता है। विभिन्न जीवों के जीनोम अनुक्रमों की बढ़ती संख्या के साथ, एक प्रमुख कदम समान जीनों की पहचान करना है जो समानता खोज और जीन एनोटेशन (कार्यात्मक मान निर्धारित करना) के माध्यम से होता है। दूसरे, कई अनुक्रमों के बीच अनुक्रम तुलनाएँ उन्नत कम्प्यूटेशनल जीनोमिक्स उपकरणों जैसे BLAST, ClustalW, Phylip द्वारा की जा सकती हैं ताकि विकासवादी संबंधों को समझा जा सके। इस क्षेत्र में तेज़ प्रगति ने संरचनात्मक जीनोमिक्स में उन्नति को जन्म दिया है जो जीनोम द्वारा कोडित प्रोटीनों की त्रि-आयामी जानकारी से संबंधित है।
शक्तिशाली कम्प्यूटर ग्राफिक्स प्रोसेसिंग यूनिट्स (GPU) और ग्राफिक कार्डों के क्षेत्र में उन्नति का उपयोग [सामान्य उद्देश्य (GP-GPU) और कम्प्यूटर यूनिफाइड डिवाइस आर्किटेक्चर (CUDA) प्रोग्रामिंग] वैज्ञानिक कम्प्यूटिंग में किया गया है जिसमें कम्प्यूटेशनल जीनोमिक्स भी शामिल है ताकि त्वरित डेटा विश्लेषण के लिए उच्च प्रदर्शन कम्प्यूटिंग लाई जा सके।
जीनोमिक्स कार्य पूंजी-गहन होता है। चूँकि जीनोमिक्स जीव विज्ञान के अध्ययन का एक प्रमुख उपकरण बन गया है और जैवप्रौद्योगिकी पर इसका गहरा प्रभाव पड़ा है, जैवप्रौद्योगिकी विभाग (विज्ञान और प्रौद्योगिकी मंत्रालय, भारत सरकार के अंतर्गत) ने भारत में तीन राष्ट्रीय जीनोमिक्स कोर सुविधाएँ स्थापित करने की पहल की है। ये सुविधाएँ भारतीय वैज्ञानिकों और उद्यमियों को अपने जीनोमिक परियोजनाओं को क्रियान्वित करने में सहायता प्रदान करेंगी। ये सुविधाएँ दो राष्ट्रीय प्रयोगशालाओं (सेंटर फॉर डीएनए फिंगरप्रिंटिंग एंड डायग्नोस्टिक्स, हैदराबाद और नेशनल इंस्टीट्यूट ऑफ बायोमेडिकल जीनोमिक्स, कल्याणी) और एक विश्वविद्यालय (इलाहाबाद विश्वविद्यालय, इलाहाबाद) के भीतर स्थित हैं।
5.5.1 संरचनात्मक जीनोमिक्स
संरचनात्मक जीनोमिक्स का अर्थ दो अलग-अलग समूहों के लिए दो अलग-अलग चीज़ें होती हैं। जो लोग प्रोटीन संरचना में रुचि रखते हैं, उनके लिए यह किसी विशेष जीनोम द्वारा एन्कोड किए गए प्रत्येक प्रोटीन की 3D संरचना को संदर्भित करता है। जो लोग गुणसूत्र संगठन में रुचि रखते हैं, उनके लिए यह संपूर्ण जीनोम की भौतिक प्रकृति को संदर्भित करता है।
पहले, संरचनात्मक जीनोमिक्स (जैसा कि संरचनात्मक जीनोमिक्स कंसोर्टियम द्वारा परिभाषित किया गया था) का केंद्रबिंदु नव-क्रमबद्ध जीनोमिक डीएनए में नए प्रोटीन फोल्ड या संरचना की खोज करना था। इस दृष्टिकोण के माध्यम से वैज्ञानिक अधिक प्रोटीन संरचनाओं को हल करने की आवश्यकता को संबोधित करते हैं, विशेष रूप से उन प्रोटीनों की जिनकी अज्ञात फोल्ड प्रकारों वाली अनुक्रम हैं।
संरचनात्मक जीनोमिक्स का विस्तृत दृष्टिकोण इस बात का अध्ययन करता है कि डीएनए क्षेत्र गुणसूत्र में और जीनोम का न्यूक्लियोसोम स्थिति कैसे संरचित है। इसमें संबंधित प्रजातियों की जीनोम संगठन में होने वाले बड़े संरचनात्मक परिवर्तनों का भी अध्ययन शामिल है। एक रोचक उदाहरण यह देखना है कि चूहे और मनुष्य में समान जीन दोनों प्रजातियों के विभिन्न गुणसूत्रों में कैसे पुनर्वितरित होते हैं।
5.5.2 कार्यात्मक जीनोमिक्स
कार्यात्मक जीनोमिक्स यह समझने का प्रयास करता है कि जीनोम में संकेतित शारीरिक कार्य की जानकारी किस प्रकार कार्यान्वित होती है। RNAseq, ChipSeq, मेटाजीनोमिक्स (सूक्ष्मजीवी समुदाय का) आदि जीनोम-आधारित उपकरणों और तकनीकों के एक समूह का उपयोग कोशिका की अवस्था से संबद्ध शारीरिक और रोगजनक कार्य को समझने के लिए किया जाता है।
5.5.3 तुलनात्मक जीनोमिक्स
तुलनात्मक जीनोमिक्स कई प्रजातियों या एक ही प्रजाति के कई व्यक्तियों के जीनों और जीनोम की तुलना से संबंधित है। यह संबंधित प्रसिद्ध जीनोम से तुलना के आधार पर नवीनतम अनुक्रमित जीनोम की व्याख्या में सहायक हो सकता है। यह उन सामान्य जीनों के समूह की पहचान करने में भी सहायक हो सकता है जो कोर जीनोम बनाते हैं और अन्य जीन जो किसी प्रजाति के लिए अद्वितीय हैं। बाद वाले किसी विशेष प्रजाति के अद्वितीय व्यवहार या कार्य के लिए उत्तरदायी हो सकते हैं। तुलनात्मक जीनोमिक्स यह भी पहचान सकता है कि कोई कार्यात्मक अणु जीनोम में उपस्थित है या अनुपस्थित है। इसे जीनोम-आधारित वर्गीकरण और वंशावली वंश अध्ययन की प्रारंभिक बिंदु के रूप में भी उपयोग किया जा सकता है।
5.6 प्रोटीन अभियांत्रिकी
इस इकाई में, हम rDNA प्रौद्योगिकी के एक और उन्नत उपयोग के बारे में सीखेंगे जिसका उपयोग इंजीनियर्ड प्रोटीन बनाने और उत्पादन करने के लिए किया जा सकता है जो मूल प्रोटीन की तुलना में अतिरिक्त या विस्तारित गुण रखते हैं।
इंजीनियर्ड प्रोटीन में ऊंचे तापमान, $\mathrm{pH}$ में परिवर्तन, लवणों या कार्बनिक विलायक की उपस्थिति के संपर्क में आने पर अधिक स्थिरता हो सकती है। प्रोटीन को अनुसंधान, निदान और चिकित्सा के लिए नए रिएजेंट विकसित और उत्पादित करने के लिए भी इंजीनियर किया जा सकता है।
5.6.1 प्रोटीन इंजीनियरिंग के अनुप्रयोग
किसी प्रोटीन को इंजीनियर करने के लिए, वांछित लक्षण के लिए उत्तरदायी महत्वपूर्ण अमीनो अम्लों की पहचान की जाती है। जीन पर इनके कोडन अनुक्रमों को बिंदु उत्परिवर्तन बनाकर संशोधित किया जाता है ताकि इसे उत्परिवर्तित किया जा सके या किसी अन्य अमीनो अम्ल से प्रतिस्थापित किया जा सके जो वांछित गतिविधि को नियंत्रित करता है। आइए कुछ उदाहरणों पर चर्चा करें।
1) डिटर्जेंट की सफाई क्रिया में सुधार के लिए पुनर्संयोजी प्रोटीन: सब्टिलिसिन एक प्रोटेओलिटिक एंजाइम है जिसे डिटर्जेंट में मिलाया जाता है ताकि गंदगी के प्रोटीन घटक को हाइड्रोलाइज किया जा सके। इससे डिटर्जेंट की सफाई क्रिया में सुधार होता है। सब्टिलिसिन की उत्प्रेरक त्रिक Ser 221, His 64 और Asp 32 है। हालांकि, ब्लीच की उपस्थिति में, Met 222 ऑक्सीकृत हो जाता है, जिससे सब्टिलिसिन की क्रिया कम हो जाती है। यदि Met 222 के लिए कोडन को Ala के लिए कोडन से बिंदु उत्परिवर्तन द्वारा साइट निर्देशित उत्परिवर्तन (एक प्रक्रिया जिससे किसी विशिष्ट स्थान पर बिंदु उत्परिवर्तन पेश किया जा सकता है) द्वारा प्रतिस्थापित किया जाता है, तो इंजीनियर्ड प्रोटीन ब्लीच की उपस्थिति में भी स्थिर रहता है (चित्र 5.9)।

चित्र 5.9 : सब्टिलिसिन में उत्प्रेरक त्रय
2) प्रोटीन शुद्धिकरण के लिए पुनःसंयोजक प्रोटीन: छह क्रमिक हिस्टिडिन अमीनो अम्लों वाला एक पेप्टाइड, जिसे 6-HisTag कहा जाता है, मध्यम क्षारीय pH पर निकल, जिंक और कोबाल्ट जैसी धातु आयनों से उच्च बंधन आसक्ति रखता है। 6-His-tag के इस गुण का उपयोग पुनःसंयोजक प्रोटीन के शुद्धिकरण के लिए किया जाता है। 6-His-tag के लिए एन्कोडिंग DNA को रुचिकर पुनःसंयोजक प्रोटीन के जीन क्रम से जोड़ा जाता है। एफिनिटी क्रोमैटोग्राफी द्वारा प्रोटीन शुद्धिकरण के दौरान, 6-His-tag युक्त पुनःसंयोजक प्रोटीन एगरोस जेल में मौजूद धातु आयनों द्वारा स्थिर किया जाएगा। अन्य सभी प्रोटीन जेल को धोकर हटा दिए जाएंगे। 6-His-tag युक्त पुनःसंयोजक प्रोटीन को तब एल्यूशन बफर का उपयोग करके निकाला जा सकता है (चित्र 5.10)।

चित्र 5.10: प्रोटीन शुद्धिकरण के लिए प्रोटीन इंजीनियरिंग। 6-His-tag युक्त इंजीनियर्ड प्रोटीन को निकल आयन कॉलम पर आसानी से और दक्षता से शुद्ध किया जाता है
3) प्रोटीन के स्थान का पता लगाने के लिए पुनःसंयोजक प्रोटीन
कोशिका में प्रोटीन: जेलीफ़िश Aequorea victoria से प्राप्त हरे फ्लोरोसेंट प्रोटीन (GFP) को इच्छित डीएनए अनुक्रम के साथ संलग्न किया जाता है। यह चिमेरिक डीएनए अब एक इंजीनियर्ड प्रोटीन के लिए कोड करता है जो यूवी प्रकाश के संपर्क में आने पर हरे फ्लोरोसेंट अणु के रूप में दिखाई देता है, इस प्रकार कोशिका के अंदर इसकी स्थिति को उजागर करता है (चित्र 5.11)।
4) पुनःसंयोजक प्रोटीन इम्यूनोटॉक्सिन के रूप में: पुनःसंयोजक इम्यूनोटॉक्सिन तर्कसंगत रूप से इंजीनियर्ड प्रोटीन एजेंट (अक्सर एंटी-कैंसर एजेंट) होते हैं जो एंटीबॉडी की चयनात्मकता को टॉक्सिन की कोशिका-विनाशक क्षमता के साथ जोड़ते हैं। इम्यूनोटॉक्सिन बनाने के लिए, कैंसर कोशिका मार्कर (कोशिका-सतह एंटीजन जो विशेष रूप से या प्रचुर मात्रा में कैंसर कोशिका पर मौजूद होते हैं) को पहचानने वाले एंटीबॉडी (सिंगल चेन एंटीबॉडी बॉक्स 2) के डीएनए कोडिंग क्षेत्र को टॉक्सिन (जैसे डिप्थीरिया टॉक्सिन या Pseudomonas का एक्सोटॉक्सिन) के डीएनए कोड के साथ संलग्न किया जाता है ताकि इम्यूनोटॉक्सिन बनाया जा सके (चित्र 5.12)। यह इंजीनियर्ड प्रोटीन अपने एंटीबॉडी भाग का उपयोग उन कोशिकाओं को चयनात्मक रूप से लक्षित करने के लिए करता है जो अपने संगत एंटीजन प्रदर्शित करती हैं, जबकि टॉक्सिन भाग उन कोशिकाओं को मारता है।
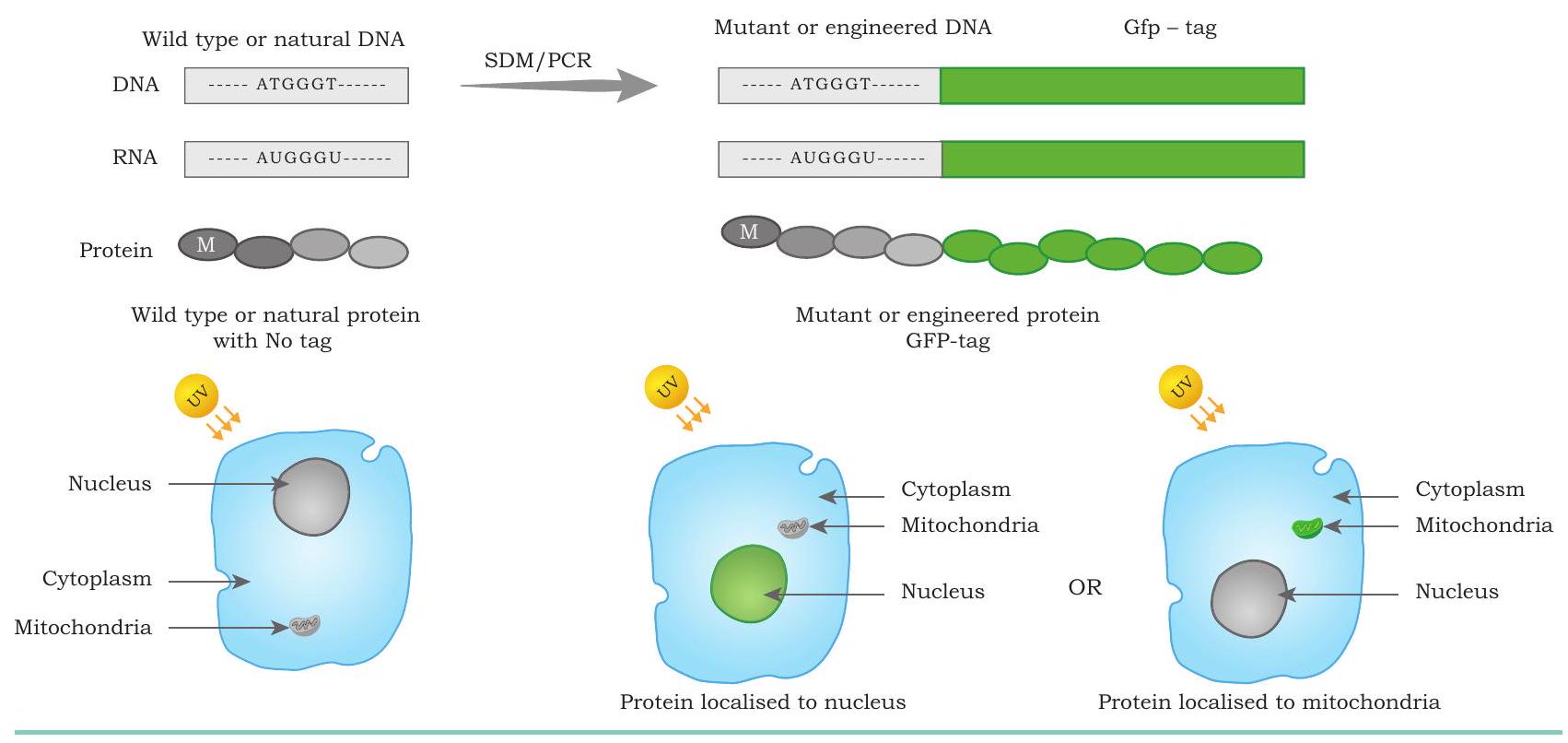
चित्र 5.11: प्रोटीन स्थानीयकरण के लिए प्रोटीन इंजीनियरिंग। ग्रीन फ्लोरोसेंट प्रोटीन (GFP) टैग के साथ इंजीनियर्ड प्रोटीन को एपीफ्लोरोसेंस या कॉन्फोकल माइक्रोस्कोपी का उपयोग कर कोशिका के विशिष्ट क्षेत्रों में आसानी से देखा और स्थानीयकृत किया जाता है। यूवी प्रकाश की उपस्थिति में नाभिक-स्थानीयकृत GFP टैग्ड प्रोटीन हरे रंग में दिखाई देता है।

चित्र 5.12: सिंगल चेन इम्यूनोटॉक्सिन। लाइट चेन कोडिंग डीएनए (Grey) का परिवर्तनशील भाग समकक्ष हेवी चेन कोडिंग डीएनए (green) और टॉक्सिन कोडिंग डीएनए (orange) से एक मनमाने लिंकर डीएनए कोड (black) के माध्यम से जुड़ा हुआ है, जिससे एक इंजीनियर्ड सिंगल चेन एंटीबॉडी जीन बनता है। इस जीन का उपयोग सिंगल चेन एंटीबॉडी बनाने के लिए किया जा सकता है। नीला त्रिकोण एक ऐसा एंटीजन है जो सिंगल चेन इम्यूनोटॉक्सिन के एंटीजन बाइंडिंग साइट पर बैठा है।
बॉक्स 2
सिंगल चेन एंटीबॉडी (SCA)
प्राकृतिक एंटीबॉडी बहु-चेन प्रोटीन होते हैं जिनमें आमतौर पर दो हेवी और दो लाइट चेन होती हैं जो दो अलग-अलग जीनों द्वारा अलग-अलग कोडित होती हैं (चित्र)। एंटीबॉडी का वह हिस्सा जो लक्ष्य एंटीजन को पहचानता है, दोनों चेनों के N-टर्मिनल क्षेत्र में स्थित होता है। एक सिंगल चेन रिकॉम्बिनेंट एंटीबॉडी में, दोनों चेनों के N-टर्मिनल भागों के DNA कोड्स को आपस में जोड़ा जाता है ताकि एक चाइमेरिक रिकॉम्बिनेंट जीन बन सके जो एक इंजीनियर्ड रिकॉम्बिनेंट प्रोटीन को कोडित करता है। यह मूल एंटीबॉडी के एंटीजन बाइंडिंग क्षेत्र को लगभग उतनी ही बंधन क्षमता के साथ बना सकता है जितनी मूल एंटीबॉडी की होती है। ऐसी सिंगल चेन एंटीबॉडीज को सुविधाजनक रूप से बैक्टीरिया में उत्पादित किया जा सकता है और एंटीबॉडी के स्थान पर थेरेप्यूटिक्स (जैसे कि रिकॉम्बिनेंट इम्यूनोटॉक्सिन के मामले में) और डायग्नोस्टिक्स (जैसे कि SCA आधारित डायग्नोस्टिक रिएजेंट्स के मामले में) में उपयोग किया जा सकता है।

सिंगल चेन एंटीबॉडी। लाइट चेन कोडित करने वाले DNA का परिवर्तनशील भाग (ग्रे) को हेवी चेन कोडित करने वाले DNA के समकक्ष भाग (हरा) के साथ एक मनमाने लिंकर DNA कोड (काला) के माध्यम से जोड़ा जाता है, जिससे एक इंजीनियर्ड सिंगल चेन एंटीबॉडी जीन बनता है जिसका उपयोग सिंगल चेन एंटीबॉडी बनाने के लिए किया जा सकता है
बॉक्स 3
मानवीकृत एकल-प्रतिरक्षी (Monoclonal) प्रतिरक्षी
एकल-प्रतिरक्षी प्रतिरक्षी अत्यंत उपयोगी अभिकर्मक होते हैं जो अपने लक्ष्यों से एक सुनिश्चित तरीके से बंधते हैं। इन प्रतिरक्षियों को आसानी से माउस हाइब्रिडोमा प्रौद्योगिकी (अध्याय 4, खंड 4.5 देखें) का उपयोग करके उत्पादित किया जाता है, जो एक जैवप्रौद्योगिकीय उपकरण है जो सामान्य माउस प्रतिरक्षा प्रतिक्रिया का लाभ उठाकर एक विशिष्ट प्रतिरक्षी उत्पादन करने वाली अमर माउस प्लाज़्मा कोशिकाएँ विकसित करता है, जिसे माउस मायलोमा कोशिकाओं से संलग्न करके बनाया जाता है। ऐसी अमर हाइब्रिड कोशिकाएँ विशिष्ट प्रतिरक्षियों के उत्पादन के लिए कोशिका कारखानों में बदल जाती हैं। यद्यपि ये माउस एकल-प्रतिरक्षी प्रतिरक्षी अभिकर्मक के रूप में उपयोगी होते हैं, इन्हें मनुष्यों में प्रतिरक्षा-चिकित्सीय रूप में उपयोग नहीं किया जा सकता। यह प्रतिबंध इसलिए है क्योंकि मानव प्रतिरक्षा प्रणाली माउस प्रतिरक्षी को विदेशी प्रतिजन के रूप में पहचानकर उसके खिलाफ प्रतिरक्षा प्रतिक्रिया उत्पन्न करती है। मनुष्यों में एकल-प्रतिरक्षी प्रतिरक्षी उत्पन्न करना व्यावहारिक नहीं है। परिणामस्वरूप, प्रोटीन अभियांत्रिकी की एक जैवप्रौद्योगिकीय रणनीति लागू की जाती है ताकि माउस एकल-प्रतिरक्षी प्रतिरक्षी को मानवीकृत किया जा सके। इस रणनीति में, माउस प्रतिरक्षी के उस भाग की पहचान की जाती है जो प्रतिजन बंधन में शामिल होता है और उसे एक मानव प्रतिरक्षी में स्थानांतरित किया जाता है, जो फिर से डीएनए स्तर पर किया जाता है। वह डीएनए कोडिंग अनुक्रम जो माउस प्रतिरक्षी के उस भाग को कोडित करता है जो प्रतिजन बंधन में शामिल होता है, को पुनः संयोजक डीएनए तकनीकों का उपयोग करके मानव प्रतिरक्षी कोडिंग जीनों में समतुल्य स्थान पर स्थानांतरित किया जाता है, जिससे माउस एकल-प्रतिरक्षी प्रतिरक्षी का एक मानवीकृत संस्करण उत्पन्न होता है (चित्र)। मानवीकृत प्रतिरक्षी को प्रायः स्वयं माना जाता है और इसलिए, चिकित्सीय उद्देश्यों के लिए प्रशासित होने पर मनुष्यों में अच्छी प्रतिरक्षा स्वीकृति होती है।

प्रोटीन अभियांत्रिकी का उपयोग करके मानवीकृत माउस Ig
सारांश
- किसी जीव का जीनोम उसका सम्पूर्ण DNA होता है, जिसमें प्रोकैरियोट्स में न्यूक्लॉयड में मौजूद DNA और यूकैरियोट्स में नाभिक, माइटोकॉन्ड्रिया और प्लास्टिड्स के गुणसूत्रों में मौजूद DNA शामिल होता है।
- जीनोम मैपिंग की दो भिन्न विधियाँ होती हैं: भौतिक मैपिंग और आनुवंशिक मैपिंग।
- एक आनुवंशिक मानचित्र जीनसूत्रों के बीच आनुवंशिक दूरी का अनुमान लगाता है, जो कि कुछ प्रसिद्ध फ़ीनोटाइप्स के लिए उत्तरदायी होते हैं। आनुवंशिक दूरी को सेंटीमोर्गन या मानचित्र इकाई में मापा जाता है (यह दो फ़ीनोटाइप्स की आनुवंशिक लिंकेज मापने की इकाई है)।
- भौतिक मैपिंग मानचित्र लक्षणों पर आधारित होती है, जैसे प्रतिबंध एंजाइम स्थल (प्रतिबंध मैपिंग) और अनुक्रम टैग किए गए स्थल (STS)।
- प्रतिबंध मैपिंग में, प्रतिबंध एंजाइम DNA को एक विशिष्ट आधार अनुक्रम पर काटते हैं, जिससे DNA के टुकड़े बनते हैं जिन्हें एगारोज जेल इलेक्ट्रोफोरेसिस द्वारा देखा जा सकता है, जिसमें DNA टुकड़ों को उनके आकार के आधार पर अलग किया जाता है।
- STS 200-500-bp के अद्वितीय DNA अनुक्रम होते हैं जिनकी स्थिति ज्ञात होती है, ये किसी जीनोम के भौतिक मानचित्र बनाने में उपयोगी लैंडमार्क के रूप में कार्य करते हैं। STS को PCR द्वारा प्रदर्शित किया जा सकता है।
- प्रथम पीढ़ी की DNA अनुक्रमण तकनीक एक बहु-चरण प्रक्रिया थी जिसमें गुणसूत्रों का पृथक्करण, प्रतिबंध पाचन और उच्च क्षमता क्लोनिंग सदिशों से लिगेशन शामिल था।
- प्रथम पीढ़ी की DNA अनुक्रमण तकनीक ने श्रृंखला समापन द्वारा अनुक्रमण की विधि का उपयोग किया, जिसमें विशिष्ट फ्लोरोसेंस टैग किए गए समापन आधारों वाले एकल सूत्री DNA की सीढ़ी को अलग किया जाता है और एक फ्लोरोसेंस डिटेक्टर द्वारा पहचाना जाता है। इस विधि द्वारा, DNA आधारों की स्थिति और प्रकार की पहचान की जाती है।
- अगली पीढ़ी की DNA अनुक्रमण तकनीक उन्नत कंप्यूटिंग एल्गोरिद्म पर निर्भर करती है ताकि लाखों छोटे अनुक्रम आउटपुट लेकर उन्हें बड़े contigs में असेंबल किया जा सके, जो बड़े जीनोमों की DNA अनुक्रमों का प्रतिनिधित्व करते हैं।
- अगली पीढ़ी अनुक्रमण में तकनीकें शामिल हैं, जैसे सम्पूर्ण जीनोम अनुक्रमण (WGS), लक्षित पुनःअनुक्रमण, नैदानिक एक्सोम अनुक्रमण आदि, और Chipseq, RNAseq अनुक्रमण तकनीकों की कुछ उन्नत अनुप्रयोग हैं।
- नैनोपोर अनुक्रमण तकनीक दो प्रोटीनों का उपयोग करती है: DNA हेलिकेज और पोरिन जैसे अणु, जो द्विसूत्री DNA को बांधते हैं, खोलते हैं और एकल सूत्री DNA को पोरिन जैसे अणुओं के माध्यम से धकेलते हैं ताकि आधार पैटर्न की पहचान की जा सके। यह तकनीक सरल, तेज, लागत-कुशल है, और परिणाम वास्तविक समय में प्रदर्शित करती है, और जीनोटाइपिंग और उच्च गतिशीलता परीक्षण के लिए उपयोगी है।
- मेटाजीनोमिक्स में किसी सूक्ष्मजीवी समुदाय में मौजूद DNA या cDNA का अनुक्रमण शामिल होता है।
- कम्प्यूटेशनल जीनोमिक्स में उच्च प्रदर्शन कंप्यूटिंग क्लस्टरों और कार्यस्थानों का उपयोग करके जीनोमिक्स डेटा का विश्लेषण शामिल होता है।
- जीनोम इंजीनियरिंग एक ऐसी तकनीक है जिससे किसी जीनोम को संशोधित किया जा सकता है (जैसे निष्क्रिय करना, हटाना, समाकलित करना, ट्रांसड्यूस करना और जीनोम को संपादित करना)।
- ट्रांसपोसोन DNA तत्व होते हैं जिनमें DNA के एक स्थान से दूसरे स्थान पर जाने की क्षमता होती है, और इनका उपयोग मौजूदा जीनों को नॉकआउट करने के लिए किया जा सकता है।
- CRISPR-Cas9 का उपयोग करके जीनोम संपादन (गाइड RNA के साथ Cas9 एंडोन्यूक्लिएस का उपयोग) डबल स्ट्रैंड ब्रेक, समजात DNA मरम्मत शामिल करता है ताकि संपादित DNA अनुक्रम उत्पन्न किया जा सके।
- संरचनात्मक जीनोमिक्स में गुणसूत्र में DNA क्षेत्र की संरचनात्मक संगठना और जीनोम के न्यूक्लियोसोम स्थिति का अध्ययन शामिल होता है।
- तुलनात्मक जीनोमिक्स उन सामान्य जीनों का समूह पहचानता है जो कोर जीनोम बनाते हैं और अन्य जीन जो किसी प्रजाति के लिए अद्वितीय होते हैं। इसलिए, यह जीनोम आधारित वर्गीकरण और विकासवादी वंशावली अध्ययन की प्रारंभिक बिंदु है।
- कार्यात्मक जीनोमिक्स का उद्देश्य कोशिका की स्थिति से संबद्ध शारीरिक और रोगजनक कार्य का अध्ययन करना है।
- प्रोटीन इंजीनियरिंग का उपयोग नए अभिकर्मक, निदान और चिकित्सा विकसित करने के लिए किया जाता है। इसका उपयोग शुद्धिकरण में आसानी के लिए 6 हिस्टिडिन-टैग (6-His-tag) युक्त पुनःसंयोजक प्रोटीन विकसित करने और प्रोटीनों की कोशिकीय स्थानीयकरण को ट्रैक करने के लिए फ्लोरोसेंट प्रोटीन विकसित करने के लिए किया जाता है।
- प्रोटीन इंजीनियरिंग तकनीक मानवीकृत मोनोक्लोनल एंटीबॉडी, सिंगल चेन एंटीबॉडी और पुनःसंयोजक इम्यूनोटॉक्सिन के उत्पादन में भी शामिल है, जो एक इंजीनियर्ड एंटीबॉडी है जो विष को विशिष्ट कोशिकीय लक्ष्यों तक पहुँचाता है।
अभ्यास
1. जीनोम क्या है? प्रोकैरियोट्स का जीनोम यूकैरियोट्स से किस प्रकार भिन्न होता है?
2. जीनोम मैपिंग के कितने प्रकार होते हैं? प्रत्येक की तुलनात्मक दृष्टिकोण से व्याख्या कीजिए।
3. मान लीजिए कि डीएनए निष्कर्षित, शुद्ध किया गया और एंजाइम बामएच1 से पचाया गया। इससे किस प्रकार की मैपिंग प्राप्त की जा सकती है? इस मैपिंग के अनुप्रयोगों का उल्लेख कीजिए।
4. एसटीएस क्या है और जीनोम मैपिंग में इसकी क्या भूमिका है?
5. डीएनए अनुक्रमण प्रौद्योगिकी और जीनोमिक कार्यप्रवाह के विकास पर संक्षिप्त अंतर्दृष्टि प्रदान कीजिए।
6. चर्चा कीजिए कि अगली पीढ़ी की डीएनए अनुक्रमण प्रौद्योगिकी ने प्रथम-पीढ़ी की डीएनए अनुक्रमण प्रौद्योगिकी की कमियों को किस प्रकार दूर किया है। विधि का विस्तार से वर्णन कीजिए।
7. भौतिक मैपिंग की इकाई क्या होती है? विस्तार से चर्चा कीजिए।
8. तीसरी पीढ़ी की अनुक्रमण प्रौद्योगिकी की विधि और अनुप्रयोगों की चर्चा कीजिए।
9. अगली पीढ़ी के अनुक्रमण के विभिन्न रूप क्या हैं? संक्षेप में समझाइए।
10. उन्नत अनुक्रमण प्रौद्योगिकियों के अनुप्रयोग क्या हैं? विस्तार से बताइए।
11. मेटाजीनोमिक्स के अनुप्रयोगों की चर्चा कीजिए।
12. जीनोम इंजीनियरिंग के लक्ष्य क्या हैं? प्रत्येक का विस्तार से वर्णन कीजिए।
13. जीनोम इंजीनियरिंग के अनुप्रयोगों में से एक उच्च स्तर की पुनःसंयोजक प्रोटीन उत्पादन है। यह हम कैसे प्राप्त कर सकते हैं? विस्तार से समझाइए।
14. जीनोम संपादन क्या है? जीनोम को संपादित करने की आवश्यकता क्यों है? क्रिस्पर-कैस9 का उपयोग कर संपादन की विधि की व्याख्या कीजिए।
15. संरचनात्मक, कार्यात्मक और तुलनात्मक जीनोमिक्स क्या हैं?
16. प्रोटीन इंजीनियरिंग के अनुप्रयोग क्या हैं? संक्षेप में चर्चा कीजिए।
17. यदि आपको 6-His-टैग वाला एक पुनः संयोजक प्रोटीन दिया जाता है, तो आप इसका उपयोग कैसे करेंगे? विधि और अनुप्रयोग को विस्तार से समझाइए।
18. प्रोटीन इंजीनियरिंग का उपयोग प्रोटीनों की कोशिकीय स्थानीयकरण को ट्रैक करने के लिए कैसे करते हैं?
19. एक पुनः संयोजक इम्यूनोटॉक्सिन कैसे उत्पन्न करते हैं? आपको क्यों लगता है कि इम्यूनोटॉक्सिन के उत्पन्न करने की आवश्यकता है? इसकी क्रिया की विधि का उल्लेख कीजिए।
20. अभिकथन: RFLP एक ही प्रतिबंधक एंडोन्यूक्लिएस द्वारा काटे जाने पर DNA खंडों की लंबाई में विचलन है।
कारण: प्रत्येक व्यक्ति के जीनोम में प्रतिबंधक स्थलों की स्थिति भिन्न होती है।
(a) दोनों अभिकथन और कारण सत्य हैं और कारण अभिकथन का सही स्पष्टीकरण है।
(b) दोनों अभिकथन और कारण सत्य हैं लेकिन कारण अभिकथन का सही स्पष्टीकरण नहीं है।
(c) अभिकथन सत्य है लेकिन कारण असत्य है।
(d) दोनों अभिकथन और कारण असत्य हैं।
21. अभिकथन: पुनः संयोजक इम्यूनोटॉक्सिन तर्कसंगत रूप से इंजीनियर्ड प्रोटीन एजेंट होते हैं।
कारण: इसे एंटीबॉडी के DNA-कोडिंग क्षेत्र को टॉक्सिन के DNA कोड से जोड़कर तैयार किया जाता है।
(a) दोनों अभिकथन और कारण सत्य हैं और कारण अभिकथन का सही स्पष्टीकरण है।
(b) दोनों अभिकथन और कारण सत्य हैं लेकिन कारण अभिकथन का सही स्पष्टीकरण नहीं है।
(c) अभिकथन सत्य है लेकिन कारण असत्य है।
(d) दोनों अभिकथन और कारण असत्य हैं।




